GALEAS™ Tumor
精準癌症基因組檢測解決方案 (NGS)
GALEAS™ Tumor 是一款先進的 NGS 檢測方案,提供全面的癌症基因組分析,涵蓋 SNV、CNV、INDEL、TMB、MSI 及關鍵免疫腫瘤學生物標誌物
先進的次世代定序腫瘤突變檢測方案
GALEAS™ Tumor 是一項領先業界的次世代定序(NGS)解決方案,專為癌症基因體分析而設計,能深入剖析腫瘤基因變異,協助偵測具臨床意義的生物標記,提供腫瘤突變負荷(TMB)、微衛星不穩定性(MSI)與結構變異等關鍵資訊。
The table of contents
GALEAS™ Tumor 特色亮點
臨床相關、內容即時更新
- 依循英國國民健康署(UK NHS)National Genomic Test Directory、NCCN、FDA 與 ESMO 最新臨床指引
- 涵蓋 519 個基因,包括 SNV、INDEL、CNV 及融合基因等
- 提供 TMB 與 MSI 評分,協助免疫療法預測
- 涵括 64 個藥物基因體 SNPs, 遺傳性與小兒腫瘤相關資訊,並支援實體腫瘤的 HLA 分型
高階免疫腫瘤學標記檢測能力
- 整合 TMB 與 MSI 分析,全面評估腫瘤基因體不穩定性
- 支援精準醫療策略,提升免疫治療預測準確性
強大易用的 GALEAS™ 分析軟體
- 雲端平台,快速且準確進行變異分析(涵蓋 SNV、INDEL、SV、CNV、TMB、MSI)
- 高解析度 CNV 偵測(>1Mb),提升結構變異辨識能力
- 友善操作介面,搭配面板共同開發的生物資訊解決方案
GALEAS™ Tumor 定序面板設計
此次世代定序(NGS)方案涵蓋關鍵驅動突變與免疫腫瘤學核心生物標記,具備以下特點:
- 身分識別追蹤用 SNPs
- 涵蓋 519 個基因 的 SNV、CNV、INDEL 分析
- 強化 CNV 背景設計,提升複製數變異(Copy Number Variation)偵測準確性
- 全面涵蓋與膠質瘤相關的 1p/9q 共缺失區域
- 可偵測結構變異,包括:ALK、BRAF、EGFR、FGFR2、FGFR3、NTRK1、NTRK2、RET、ROS1、TMPRSS2
- 收錄 64 個與腫瘤學相關的藥物基因體 SNPs
- 提供實體腫瘤特性分析所需的 HLA 基因分型資料
高精度變異檢測能力
SNV 與 INDEL 準確度
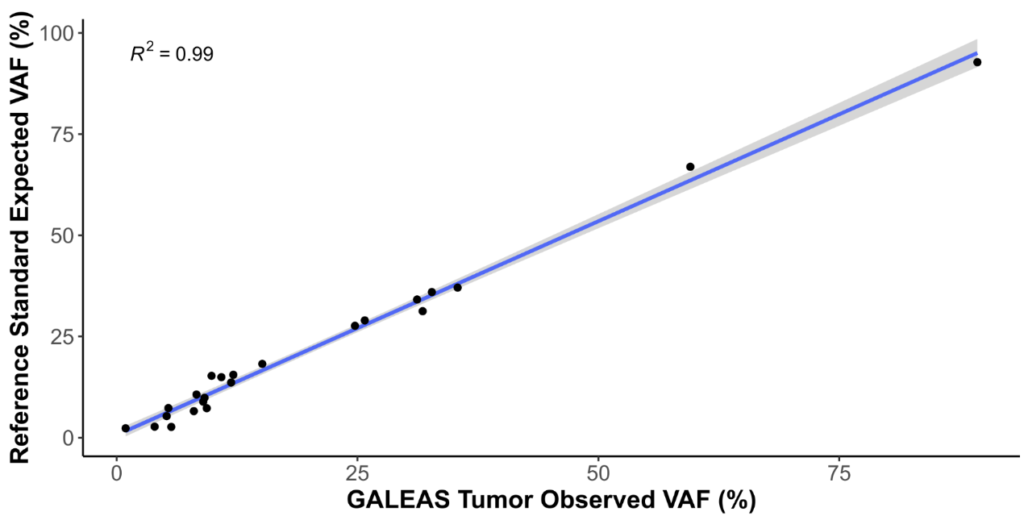
- 透過 FFPE 參考樣本進行驗證,在 500x 深度下達成 R² 相關係數 0.99(圖1)
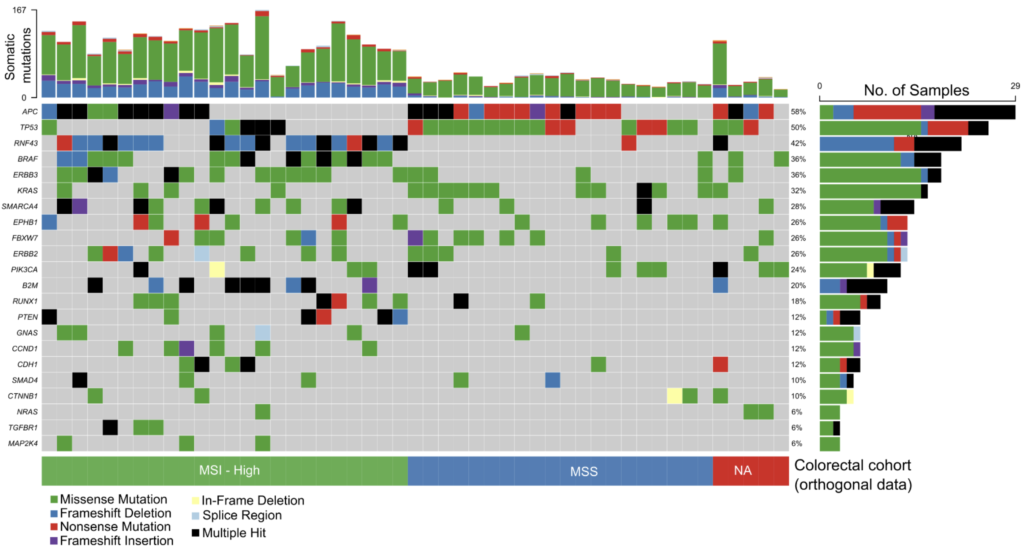
- 在 50 個大腸直腸癌(CRC)樣本中達成 100% 體細胞突變檢出準確率(圖2)
複製數變異(CNV)檢測
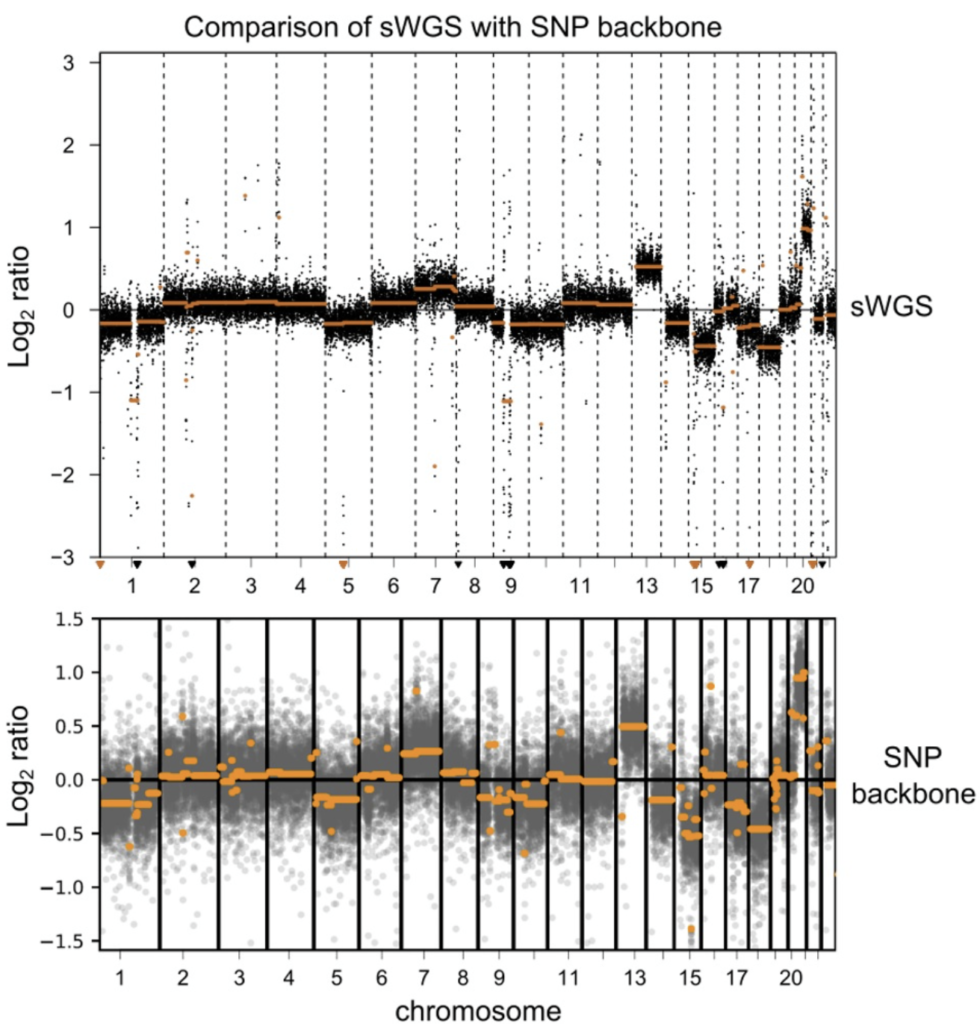
- 提供解析度高於 1Mb 的 CNV 偵測能力
- GALEAS™ Tumor CNV 數據與淺層全基因體定序(sWGS)結果呈現高度相關性(圖3)
- 成功驗證 EGFR 與 MET 基因的 CNV 變異偵測能力(分別測試 3、6、12 拷貝數)
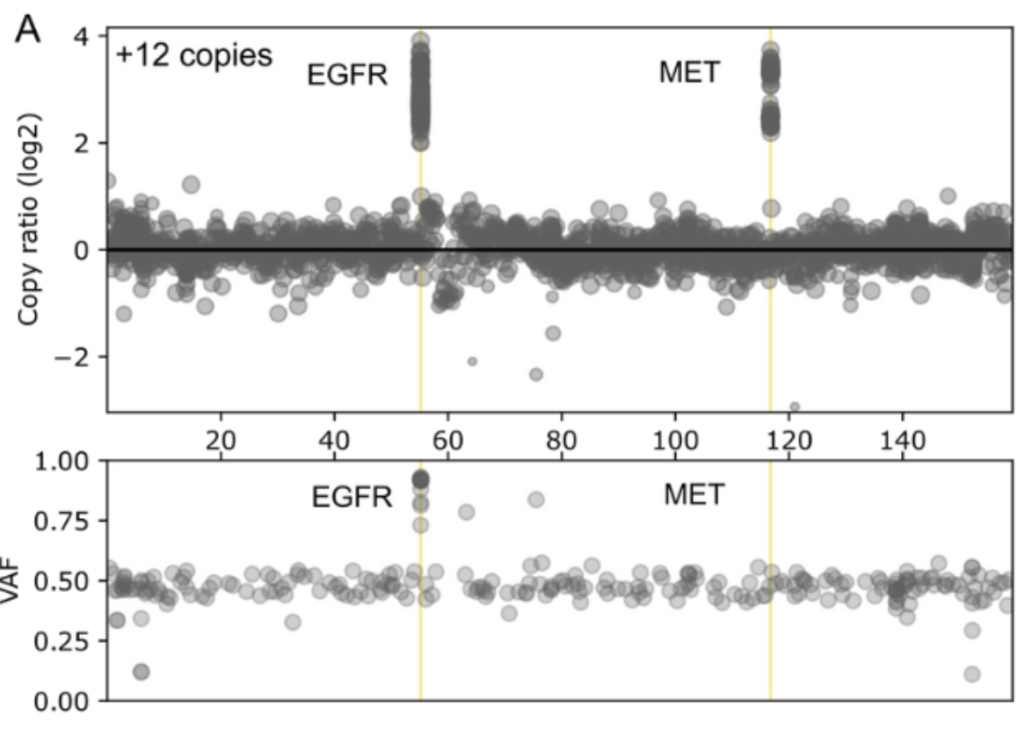
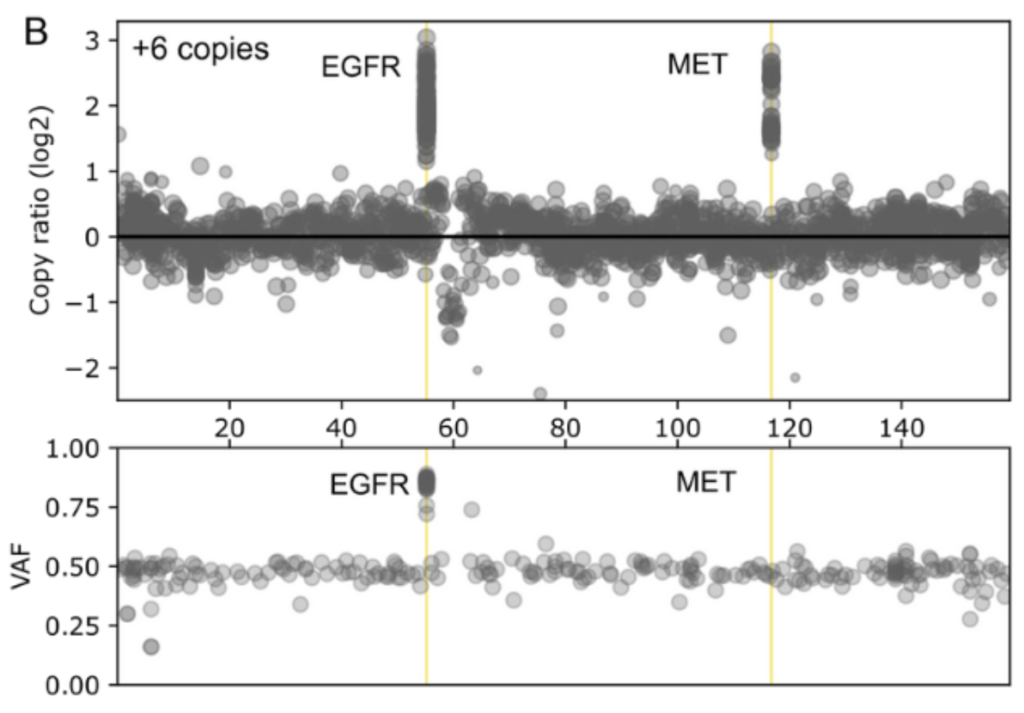
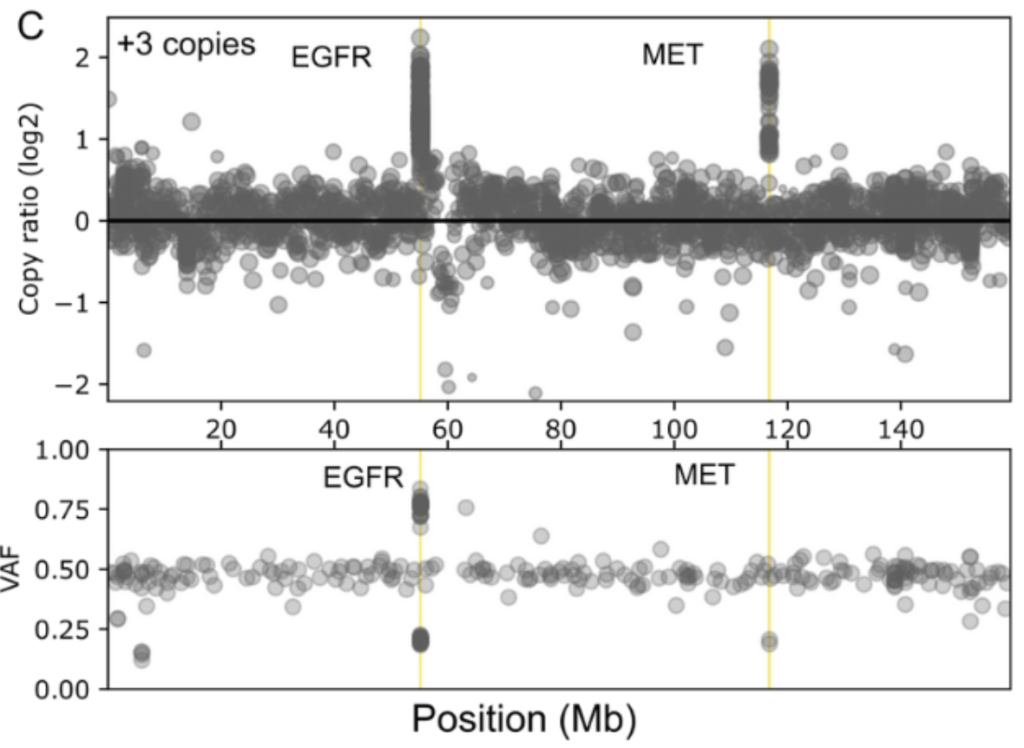
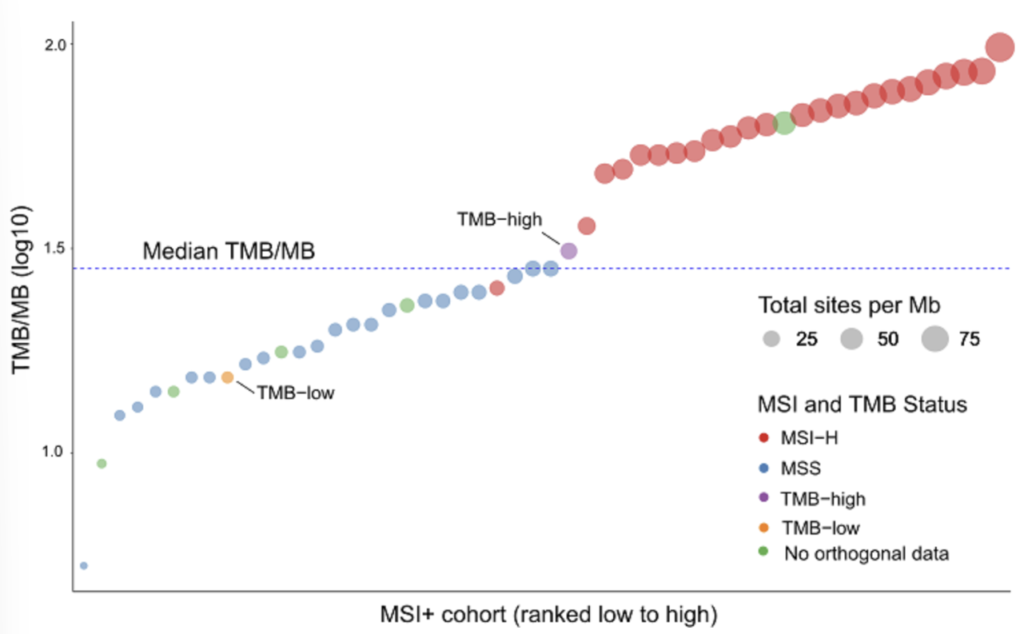
微衛星不穩定性(MSI)評分
- 在 24 個 CRC FFPE 樣本中成功檢出 23 個 MSI-High 狀態
- 可精準區分正常樣本、微衛星穩定(MSS)及高度不穩定(MSI-H)樣本
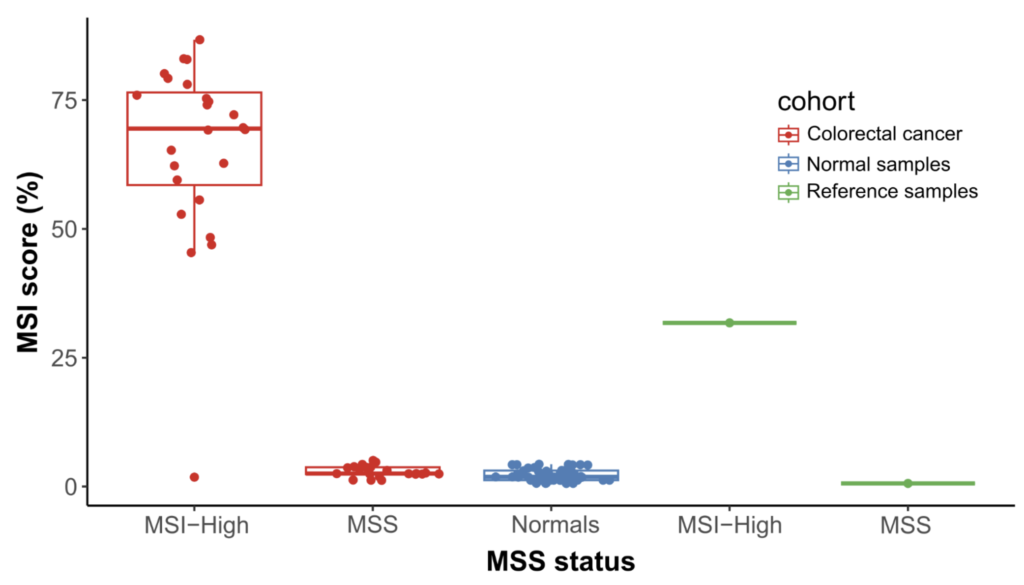
腫瘤突變負荷(TMB)分析
- 在 CRC 樣本中 TMB 分數與 MSI 狀態呈現高度相關性
- 即使在低突變量的小型定序面板中,亦能提供穩定可靠的 TMB 評分
- 高目標區域擷取率,大幅提升定序效率
- 在臨床關鍵基因上實現均一覆蓋,確保數據一致性
- 支援精準且具重現性的變異檢出,提升臨床應用可靠性
GALEAS™ 分析軟體
- 專為提升 GALEAS™ Tumor 面板效能 而設計
- 支援 準確的 TMB/MSI 分析,並結合精簡高效的生物資訊分析流程
GALEAS™ Tumor 是一套經臨床驗證的全方位次世代定序(NGS)解決方案,可在單一工作流程中完成 519 個基因的 SNV、CNV、INDEL 及 TMB/MSI 分析。
藉由強化的探針設計、全面的基因覆蓋範圍與高度一致的覆蓋深度,GALEAS™ Tumor 可精準且靈敏地檢出 SNV、INDEL、SV 及 CNV。當搭配 GALEAS™ Analysis Software 使用時,從樣本準備到分析,皆能提供直覺化且友善的使用流程。
References
- Ciriello G, Miller ML, Aksoy BA, Senbabaoglu Y, Schultz N, Sander C. Emerging landscape of oncogenic signatures across human cancers. Nature genetics. 2013;10):1127-33.
- The ICGC/TCGA PanCancer Analysis of Whole Genomes Consortium. Pan-cancer analysis of whole genomes. Na- ture. 2020: 82-93.
- Endris V, Buchhalter I, Allgäuer M, Rempel E, Lier A, Vol- ckmar AL, et al. Measurement of tumor mutational bur- den (TMB) in routine molecular diagnostics: in silico and real-life analysis of three larger gene panels. International journal of cancer. 2019;144(9):2303-12.
- Schrock AB, Ouyang C, Sandhu J, Sokol E, Jin D, Ross JS, et al. Tumor mutational burden is predictive of response to immune checkpoint inhibitors in MSI-high metastatic colorectal cancer. Annals of Oncology. 2019;30(7):1096-103.
產品資訊
| Parameters | Specification |
|---|---|
| Enrichment method | Hybridization and capture |
| Number of genes | 519 |
| Capture Panel Size | 3.74Mb |
| Sequencing platform | Illumina |
| Targets | Genes associated with common cancers |
| Variant types | SNVs, CNVs and indels |
| Input DNA requirements | 10ng-200ng |
| Sample type | gDNA from FFPE, frozen tissue or blood |
| Multiplexing guidance for sequencing | 25M reads per sample required to achieve 500x. This equates to 5Gb per sample |
訂購資訊
| Product Name | Catalogue No. |
|---|---|
| GALEAS Tumor Kit Frag A (96 samples) | NGS_GAL_TCP_FR_96_A |
| GALEAS Tumor Kit Frag B (96 samples) | NGS_GAL_TCP_FR_96_B |
| GALEAS Tumor Kit Frag C (96 samples) | NGS_GAL_TCP_FR_96_C |
| GALEAS Tumor Kit Frag D (96 samples) | NGS_GAL_TCP_FR_96_D |
| GALEAS Tumor Kit Frag (16 samples) | NGS_GAL_TCP_FR_16 |
- A: Adapter標號 1~96
- B: Adapter標號 97~192
- C: Adapter標號 193~288
- D: Adapter標號 289~384