GALEAS™ HereditaryPLUS
專為精準腫瘤學設計的遺傳性腫瘤基因檢測方案
GALEAS™ HereditaryPLUS 涵蓋146個與遺傳性腫瘤相關的基因,精準偵測SNV、INDEL、CNV及嵌合變異,為癌症風險評估提供強大支持。
遺傳性癌症風險的先進基因檢測方案
GALEAS™ HereditaryPLUS 是一款領先的遺傳性腫瘤檢測面板,專為精準評估遺傳癌症變異而設計。此面板涵蓋 146個臨床相關基因,與乳癌、前列腺癌、林奇症候群、威爾姆氏腫瘤等多種遺傳性癌症密切相關。
The table of contents
GALEAS™ HereditaryPLUS 的關鍵特色
全面性的基因覆蓋
- 鎖定與遺傳性腫瘤相關的 146個高風險基因
- 涵蓋常見癌症類型,如乳癌、前列腺癌
- 包含罕見腫瘤類型,如嗜鉻細胞瘤(pheochromocytoma)與威爾姆氏腫瘤(Wilms tumor)
- 涵蓋具臨床意義的非編碼區域(例如:BRCA1/2 的5′ UTR與APC啟動子)
表一:GALEAS™ HereditaryPLUS 所涵蓋之與遺傳性癌症風險相關,並被臨床指引納入的基因列表
| Cancer type | Recommended genes for screening included in GALEASTM HereditaryPlus |
|---|---|
| Breast | ATM, BARD1, BRCA1, BRCA2, CDH1, CHEK2, NBN, NF1, PALB2, PTEN, STK11, TP53 |
| Colon | TAPC, AXIN2, BMPR1A, CHEK2, EPCAM, GREM1, MLH1, MSH2, MSH6, PMS2, MSH3, MUTYH, NTLH1, POLD1, POLE, PTEN, RNF43, SMAD4, STK11, TP53 |
| Renal | BAP1, FH, FLCN, MET, SDHB, VHL |
| Ovarian | ATM, BARD1, BRCA1, BRCA2, CDH1, CHEK2, NBN, NF1, PALB2, PTEN, SKT11, TP53, RAD51C, RAD51D |
| Prostate | ATM, BRCA1, BRCA2, CHEK2, MLH1, MSH2, MSH6, PALB2 |
| Gastric/GIST | CDH1, KIT, PDGFRA, SDHC, SDHD, SDHA |
| Brain | APC, ATM, MLH1, MSH2, MSH6, PMS2, TP53 |
| Sarcoma | EXT1, EXT2, MTAP, NF1, RECQL4, SQSTM1, TP53 |
| Paediatric* | CDKN1C, CTR9, REST, TRIM28, WT1 |
優異的準確性與重現性
- 先進探針設計,精準偵測SNV、INDEL與CNV變異
- 高均一性與完整覆蓋範圍,提升診斷可靠性
- 使用標準對照樣本 NA24385 驗證437個SNP位點
表二:GALEAS™ HereditaryPLUS 在NA24385標準樣本中之SNV與INDEL重現性分析(4次重複實驗)
| Recall | |
|---|---|
| SNV | 99.78% |
| Indel | 100% |
GALEAS™ 專屬分析軟體 — 優化生物資訊分析流程
- 基於雲端的分析流程,提供高準確率的變異判讀
- 內建「正常樣本資料庫(Panel of Normals)」機制,提升CNV偵測準確度
- 支援關鍵基因(如 APC、TSC2)中嵌合型CNV(Mosaic CNV)之偵測
精準偵測複製數變異(CNVs)
- 於NIBSC林奇症候群 MLPA 細胞株中達成 100% 準確率
- 可重現性高,支援偵測單一外顯子至全基因範圍的CNV
- 有效偵測 嵌合型CNV,適用於重要癌症風險基因
表三:使用 GALEAS™ HereditaryPLUS 偵測 NIBSC 參考對照組中 CNV 的召回率與精確度統計
| CNV | Genotypic sex | CNV type | Detected |
|---|---|---|---|
| Copy normal | Male | Copy neutral | Yes |
| MSH2 deletion exons 1-6, heterozygous | Male | Multi-exon deletion | Yes |
| MSH2 deletion exon 7, heterozygous | 1 Male | Single exon deletion | Yes |
| MSH2 deletion exons 1-2, heterozygous | female | Multi-exon deletion | Yes |
| MSH2 deletion, exon 1, heterozygous | Male | Single exon deletion | Yes |
| MLH1 exon 13 amplification (3 or more copies) | Female | Multi-exon amplification | Yes |
優異的測序表現
- 高目標區讀取率、低重複率、均勻的覆蓋深度
- 99%目標區域覆蓋率達30x以上,確保偵測準確性
- 相較於他牌產品,GALEAS™ 提供更多內容(含CNV探針),而測序成本僅增加不到10%
表四:GALEAS™ HereditaryPLUS 與市售他牌面板之測序效能比較
| Key Quality Indicator | GALEAS™ Hereditary Plus | Company I |
|---|---|---|
| Number of genes | 146 | 113 |
| Capture panel size (kb) | 809 kb | 403 kb |
| GB required for mean 100x coverage | 0.2 Gb | 0.12 Gb |
| Percentage coverage >30x | 99% | 96% |
| Percentage on or near bait | 81% | 61.51% |
| Percent duplication | 2.0% | 8.99% |
| SNV recall | 99.7% | 98.1% |
| INDELs recall | 100% | 97.2% |
臨床驗證數據
SNV偵測準確率
- 在臨床樣本中達成 SNV召回率100%
- 成功偵測多樣變異類型,包括 大於10 bp的INDEL
表五:GALEAS™ HereditaryPLUS 於臨床樣本中之SNV召回率統計
| ID | Gene | HGVS coding | HGVS protein | Genomic position |
|---|---|---|---|---|
| 22 | BRCA1 | c.1175_1214del | p.Leu392fs*5 | chr17:43094317 |
| 23 | BRCA1 | c.1175_1214del | p.Leu392fs*5 | chr17:43094317 |
| 64 | MSH2 | c.942+3A>T | P.? | chr2:47414421 |
| 65 | PMS2 | c.736_741delins TGTGTGTGAAG | p.(Pro246Cysfs*3) | chr7:5997389 |
| 66 | MLH1 | c.1946dupC | p.(Leu650Phefs*14) | chr3:37048561 |
| 67 | MSH2 | c.1213_1217dup | p.(Leu407Thrfs*7) | chr2:47429877 |
| 68 | MSH6 | c.3562_3563del | 1 p.(Ser1188Tyrfs*5) | chr2:47805623 |
複製數變異(CNVs)偵測能力
結合 GALEAS™ HereditaryPLUS 面板 與 GALEAS™ 分析軟體,可提供:
- 精準偵測 PMS2 複製缺失變異(圖1C)
- 可涵蓋從單一外顯子至整個基因範圍的CNV
- 可偵測如 APC 與 TSC2 等關鍵基因的 嵌合型CNV (圖1E,1F)
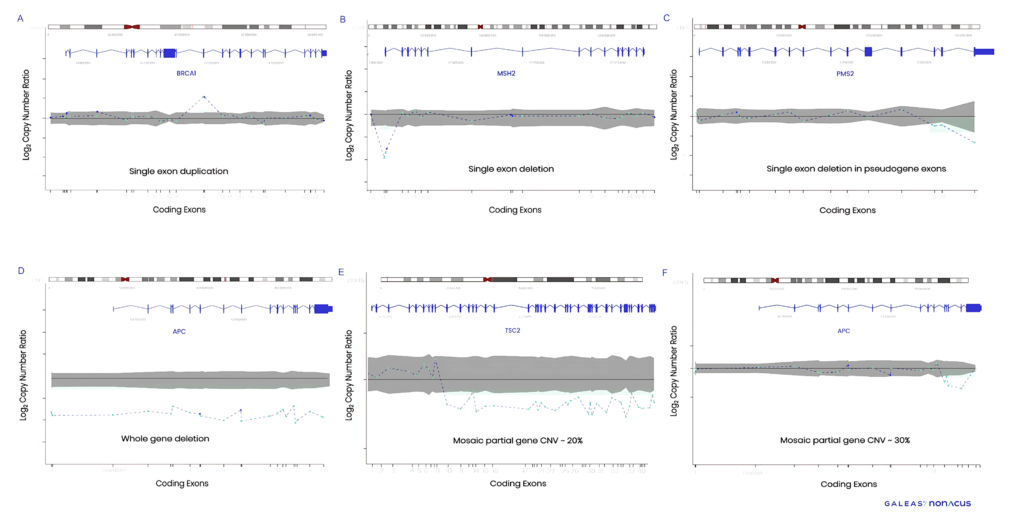
A)BRCA1 單一外顯子重複, B)MSH2 單一外顯子缺失, C)PMS2 假基因多外顯子缺失, D)APC 整個基因缺失, E)TSC2 部分基因嵌合型CNV(-20%),F)APC 部分基因嵌合型CNV(-30%)
參考文獻
1. Ngeow J, Eng C. Precision medicine in heritable cancer: when somatic tumour testing and germline mutations meet. NPJ Genomic Medicine. 2016;(1):1-3.
產品資訊
| Parameters | Specification |
|---|---|
| Enrichment method | Hybridisation and capture |
| Number of genes | 146 |
| Capture Panel Size | 809 Kb |
| Sequencing platform | Illumina |
| Targets | Genes associated with hereditary cancer |
| Variant types | SNVs, INDELs and CNVs |
| Sample type | gDNA from blood or saliva |
| Input DNA requirements | 10-200 ng |
| Multiplexing guidance for sequencing | 1 million reads per sample required to achieve 100×. This equates to 0.2 Gb per sample |
訂購資訊
| Product | Catalog No. |
|---|---|
| GALEAS™ HereditaryPlus – 16 samples (with enzymatic fragmentation for gDNA or FFPE) | NGS_GAL_HCP_FR_16 |
| GALEAS™ HereditaryPlus – 96 samples (with enzymatic fragmentation for gDNA or FFPE) | NGS_GAL_HCP_FR_96_A/B/C/D* |
- A: Adapter標號 1~96
- B: Adapter標號 97~192
- C: Adapter標號 193~288
- D: Adapter標號 289~384
* 為了提供大量樣本檢測的靈活性,我們在檢體試劑盒中附有四款Adapter
我們的產品
nonacus
NGS相關解決方案
相關應用_以變異類型分類
相關解決方案_以疾病類別分類
癌症基因分析解決方案
生殖細胞變異分析(germline mutations)
遺傳性疾病
GALEAS™系列
Products
nonacus
NGS Workflow Solutions
Mutation Analysis Applications
Disease-Specific Applications
Cancer Genomic Analysis Solutions
Germline Variant Analysis
Hereditary Diseases
GALEAS™ Series