Cell3™ Target: Nexome
精確變異檢測的全面外顯子捕獲解決方案
Cell3™ Target: Nexome 提供增強型外顯子捕獲,精確檢測 SNV、INDEL 和 CNV,實現更廣泛的變異捕捉、優化效率及可靠的臨床見解
無與倫比的基因洞察,精確的 SNV、INDEL 和 CNV 檢測
體驗精確的 SNV、INDEL 和 CNV 檢測於單一檢測中
Cell3™ Target: Nexome 超越了傳統全外顯子定序基因檢測(WES),針對基因組中不僅是蛋白質編碼區域,還包括臨床相關的非編碼區域。此外顯子捕捉技術經過強化探針設計,實現高信心的 CNV 檢測,確保全面的基因組分析。
主要特點
- 廣泛覆蓋
-
針對 51.9 Mb 的基因組,超越傳統外顯子面板的覆蓋範圍
- 準確的變異檢測
-
針對 SNV、INDEL 和 CNV 進行優化,具有卓越的回憶率
- 臨床相關增強功能
-
包含與產前診斷、癲癇和藥物基因組學(PGx)相關的基因
- 效能優化
-
在不增加定序成本的情況下,能夠檢測 30% 更多的變異
- 可自動化工作流程
-
精簡的流程,支持高通量處理,最多可處理 96 份樣本
臨床應用的全面基因洞察
- 通常可通過 MLPA 測試檢測到的外顯子級別的缺失和重複
- 與產前異常相關的基因,有助於產前診斷
- 幼兒癲癇性腦病(EIEE)轉錄本,增強癲癇診斷
- 針對 OMIM 病理基因集的 4,090 個基因的 RefSeq 轉錄本、啟動子區域、5′ 和 3′ UTR 序列
- 與遺傳性疾病相關的非編碼變異,提供更廣泛的分析範圍
- 藥物基因組學(PGx)標記,提升藥物反應預測能力
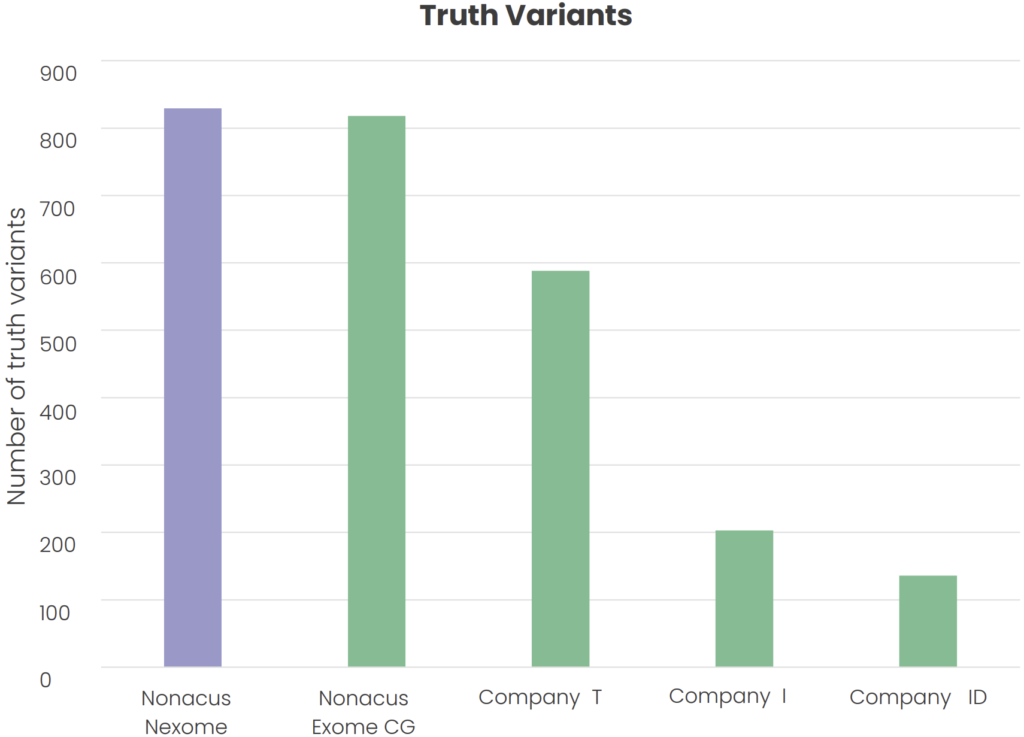
無與倫比的 SNV 和 INDEL 檢測精度
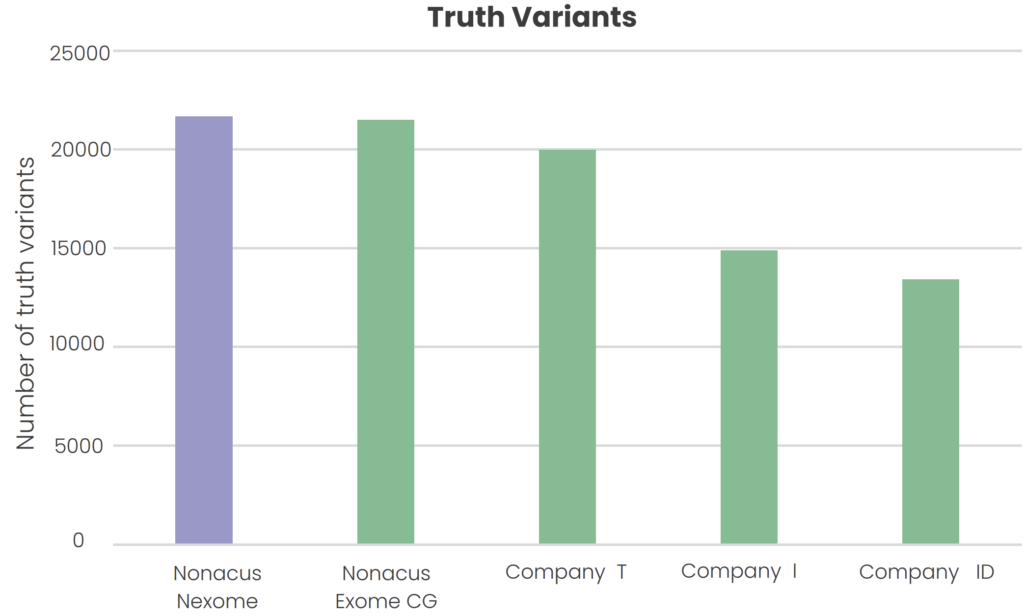
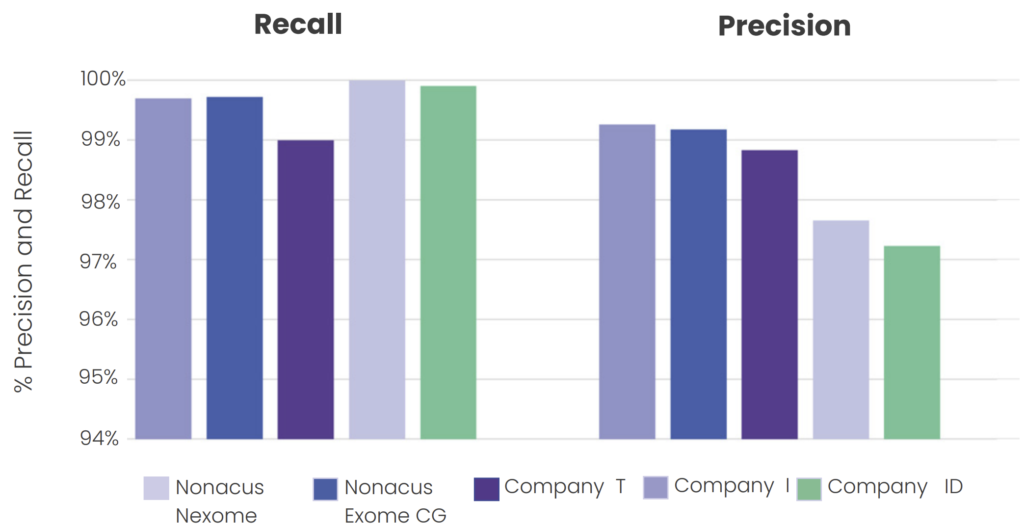
Cell3™ Target: Nexome 在檢測 HG001 人類基因組標準參考中的真實變異方面超越了競爭對手的外顯子面板,確保卓越的準確性和回憶率。
- 更高的真實變異檢測率
穩定地識別出比領先競爭對手更多的 SNV 和 INDEL。 - 維持卓越的精準度
對臨床相關突變提供高準確度。
SNV

INDELS
具成本效益且高產量的全外顯子定序基因檢測(WES)
更廣泛的變異捕捉
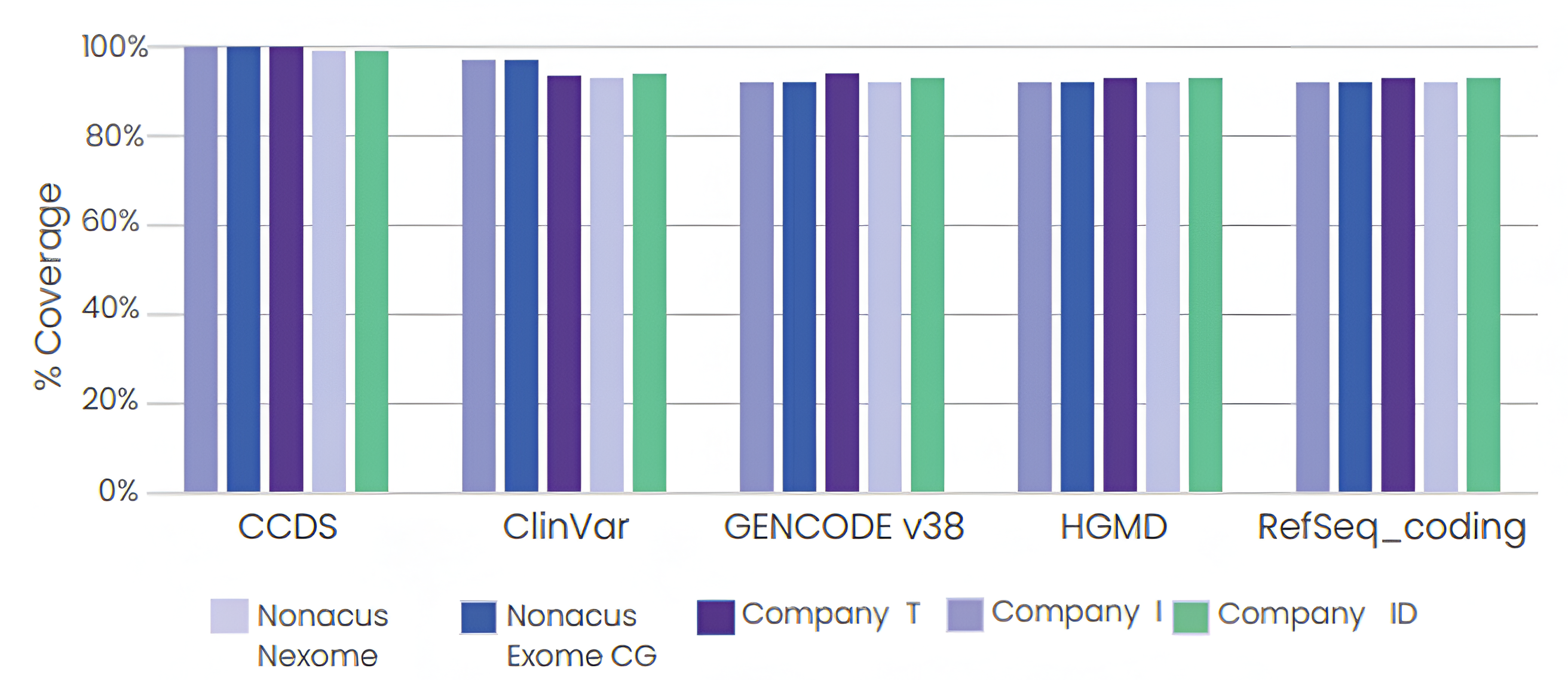
Cell3™ Target: Nexome 最大化定序效率,只需 6.63 Gb 即可實現 100× 的平均覆蓋度,降低不必要的定序成本。
- 比傳統外顯子試劑盒檢測多 30% 的變異。
- 針對超過 51 Mb 的人類基因組進行定序。
表1 Cell3™ Target: Nexome 實現 100× 平均覆蓋所需的 Mb。
| Panel Size (Mb) | Percentage target covered at 1x | Gb Required for mean 100x coverage | Percent Bases on or near bait | |
|---|---|---|---|---|
| Nexome | 51.90 | 98.78% | 6.63 | 94.18% |
| Exome CG | 51.60 | 98.78% | 6.57 | 94.07% |
| Company T | 36.70 | 97.42% | 6.85 | 85.89% |
| Company I | 45.20 | 98.15% | 7.16 | 86.18% |
| Company I.D | 34.10 | 98.49% | 6.04 | 93.09% |
可靠的 CNV 檢出
拷貝數變異(CNV)約佔疾病相關變異的 10%,並且在約 10-20% 的神經發育障礙個體中被識別。
- 覆蓋範圍從 100 bp 到 40 Mb,能自信地檢測外顯子和基因層級的 CNV
- 通過 MLPA 和 CMA 確認的 CNV 突變進行評估,從 50 bp(單個外顯子)到 42 Mb
- 臨床相關事件的檢測達到了卓越的精確度和回憶率,為 CMA 和 MLPA 基於 CNV 的分析提供了外顯子替代方案
表2a MLPA 確認的 CNV 檢測
| Affected Gene | CNV region | CNV size (bp) | CNV exons | CNV type | Bayes factor |
|---|---|---|---|---|---|
| FBN1 | exons 29-65 | 74632 | 37 | deletion | 320.0 |
| BRCA1 | exons 1-23 | 77841 | 24 | deletion | 190.0 |
| FBN1 | exons 1-17 | 142063 | 18 | deletion | 300.0 |
| BRCA1 | exons 1-17 | 57876 | 18 | deletion | 200.0 |
| BRCA1 | exons 8-13 | 17956 | 6 | deletion | 40.4 |
| BRCA1 | exons 8-13 | 17956 | 6 | deletion | 82.4 |
| BRCA2 | exons 5-7 | 513 | 3 | deletion | 22.1 |
| NSD1 | exons 7-9 | 6034 | 3 | deletion | 34.5 |
| FBN1 | exons 60-62 | 3934 | 3 | deletion | 32.8 |
| NSD1 | exons 1-3 | 58095 | 3 | deletion | 54.8 |
| BRCA2 | exons 1-2 | 1054 | 2 | deletion | 28.3 |
| BRCA1 | exons 7-8 | 311 | 2 | deletion | 4.7 |
| BRCA1 | exons 8-9 | 1444 | 2 | deletion | 7.5 |
| BRCA1 | exon 16 | 211 | 1 | deletion | 14.5 |
| BRCA1 | exon 20 | 84 | 1 | deletion | 9.4 |
表2b CMA 確認的多基因 CNV 檢測
| CNV region | CNV size (Mb) | CNV genes | CNV type | Bayes factor |
|---|---|---|---|---|
| 13q14.2q32.1 | 42.0 | 367 | deletion | 2410 |
| 4p16.3p15.2 | 22.9 | 339 | deletion | 4620 |
| 20q11.22q13.12 | 11.3 | 244 | deletion | 7000 |
| 7p14.1p11.2 | 15.9 | 182 | deletion | 5040 |
| 1p36.32 | 3.7 | 140 | deletion | 2710 |
| 22q11.21 | 2.0 | 83 | deletion | 2890 |
| 8q23.1q24.12 | 11.8 | 71 | deletion | 1330 |
| 22q11.21 | 2.2 | 64 | duplication | 1430 |
| 11p12p11.2 | 2.3 | 54 | deletion | 1240 |
| 7q11.23 | 1.4 | 38 | deletion | 2080 |
| 15q11.2 | 0.9 | 31 | deletion | 494 |
| 17p12 | 1.3 | 24 | deletion | 275 |
| 14q22.1 | 0.7 | 20 | deletion | 508 |
| 15q11.2 | 0.5 | 4 | duplication | 370 |
| 13q12.11 | 0.2 | 2 | deletion | 75 |
無縫自動化與可擴展實驗室處理
為提高效率與靈活性而設計,Cell3™ Target: Nexome 簡化了定序工作流程
- Cell3™ Target: Nexome 試劑盒包含所有必要的試劑,用於文庫準備、雜交和捕捉
- 總工作流程時間
少於 10 小時(操作時間少於 2 小時) - 最小 DNA 輸入要求
僅需 1 ng 的 DNA - 準備自動化
支持手動和高通量工作流程(最多可處理 384 份樣本) - 兼容性
完全支持所有 Illumina 定序平台的擴展
References
- McKusick V. Online Mendelian Inheritance in Man, OMIM™. McKusick-Nathans Institute for Genetic Medicine, Johns Hopkins University (Baltimore, MD) and National Center for Biotechnology Information, National Library of Medicine (Bethesda, MD), 2000. Accessed October 10, 2023. https://omim. org. 2009. Full article
- Smedley D, Schubach M, Jacobsen JO, Köhler S, Zemojtel T, Spielmann M, et al. A whole-genome analysis framework for effective identification of pathogenic regulatory variants in Mendelian disease. The American Journal of Human Genetics. 2016; 99(3):595-606. Full article
- Landrum MJ, Lee JM, Benson M, Brown GR, Chao C, Chitipiralla S, et al. ClinVar: improving access to variant interpretations and supporting evidence. Nucleic Acids Research. 2018;46(D1):D1062-7.. Full article
產品資訊
| Enrichment method | Hybridisation and capture |
| Capture panel Size | 51.9 Mb |
| Sequencing platform | Illumina |
| Targets | Clinically relevant genes |
| Variant types | SNVs, indels and CNVs |
| Sample type | gDNA from blood, saliva, amniotic fluid, tissue or FFPE, cfDNA |
| Input DNA requirements | 1-1000 ng |
| Expected percentage duplication | ~5-6% |
| Expected percentage on target (150 bp padding) | ~95% |
| Gb required for mean 100× coverage | 6.63 |
| Multiplex capability | 384 |
訂購資訊
Cell3™ Target試劑組,提供兩種建庫試劑版本。
- Fragmentation:適用於DNA樣本(Genomic、FF、FFPE)
- Non-fragmentation:適用於cfDNA樣本
| Product | Catalog No. |
|---|---|
| Cell3™ Target: Nexome, Frag 16 samples | NGS_C3T_NEX_FR_16 |
| Cell3™ Target: Nexome, Frag 96 samples | NGS_C3T_NEX_FR_96_A/B/C/D* |
| Cell3™ Target: Nexome, Non Frag 16 samples | NGS_C3T_NEX_NF_16 |
| Cell3™ Target: Nexome, Non Frag 96 samples | NGS_C3T_NEX_NF_96_A/B/C/D* |
- A: Adapter標號 1~96
- B: Adapter標號 97~192
- C: Adapter標號 193~288
- D: Adapter標號 289~384
* 為了提供樣本多重檢測的靈活性,我們在檢體試劑盒中附有四款Adapter