基因檢測技術在過去幾十年間有了革命性的進展,從早期耗時繁瑣的人工方法,演變至今日快速、準確且具成本效益的解決方案。當中,次世代定序技術(NGS),無疑是現代基因診斷與基因體研究的核心技術。但這場科技演進是如何發生的?
從桑格定序到NGS:基因科技的重大飛躍
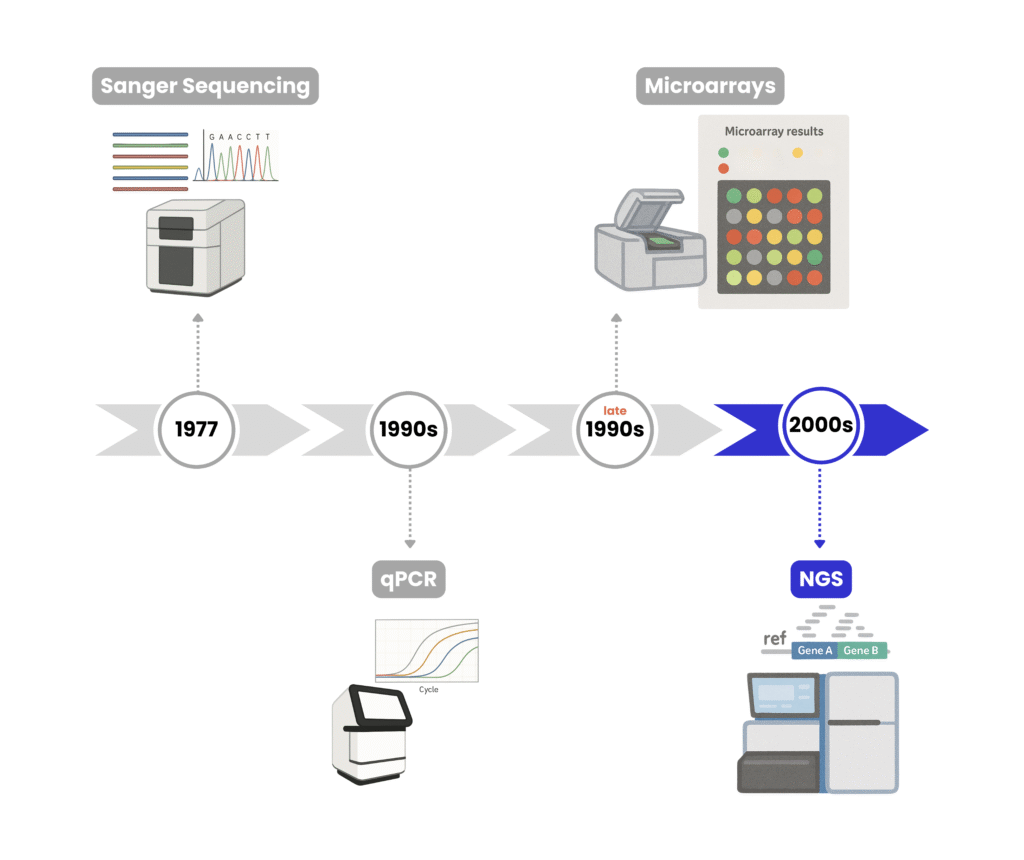
桑格定序
(Sanger Sequencing)
1977
仔細閱讀每一段文字

- 由諾貝爾獎得主Frederick Sanger開發,此技術成為DNA定序的黃金標
- 它透過合成特定核苷酸終止的DNA片段,並利用螢光標記協助判讀序列
桑格定序在「人類基因組計畫」中扮演了關鍵角色,為未來的重大突破奠定了基礎。
低通量、費時、成本高,解碼一組人類基因組需花費數年與數十億美元
即時定量PCR(qPCR)
1990年代
像是使用螢光筆搜尋特定詞彙

- 此技術著重於即時偵測與定量DNA/RNA、廣泛應用於病毒檢測與癌症基因表現研究
- COVID-19、HIV等病原體偵測、癌症研究中的基因表現分析
基因表現研究、病原體偵測
僅限目標區域分析、無法獲得序列資訊
基因晶片(Microarray)
1990 年代末期
像是掃描整本書中的關鍵詞

- 將數千個已知DNA探針固定於晶片上、能同時偵測上萬個基因變異
- 染色體變異與疾病關聯研究、發展障礙檢測(如CNV)
讓科學家能夠一次篩檢數千種基因變異、徹底革新了基因關聯性研究與大型染色體變異的診斷方式
無法偵測微小變異或未知序列
次世代定序(NGS)
2000年代至今
將整本書數位化並用拼寫檢查器快速掃描

- NGS可同時平行處理數百萬個DNA片段、極大提升速度與資料量、並大幅降低成本
讓全基因體定序在數天內即可完成、且成本僅為過去的一小部分。這項技術驅動了個人化醫療、癌症基因體學、罕見疾病診斷、甚至是傳染病疫情追蹤等領域的重大突破
高通量定序可同時讀取數十億個DNA鹼基
大幅降低長期成本、同時提升定序速度
技術比較:通量、速度與解析力
| 技術 | 主要用途 | 每次輸出量 | 定序全人類基因所需時間 |
| Sanger | 小規模目標定序 | 約1,000個鹼基 | 數年 |
| Microarray | 掃描已知變異 | 數十萬筆數據 | 不適用 |
| qPCR | DNA/RNA定量(非定序) | 1–100個基因 | 幾分鐘至數小時 |
| NGS | 全基因體分析 | 數十億鹼基 | 1-2天 |
臨床與研究應用場景
- 桑格定序:傳統定序金標準
-

- BRCA1/2等遺傳癌症基因確認
- 合成生物學中質體驗證
- 物種鑑定與系統發育研究
- qPCR:即時定量、快速偵測
-

- 新冠病毒、流感等即時病原檢測
- 癌症相關基因表現分析
- 生物製劑與環境樣本中的微生物監控
- 基因晶片:高通量快速篩查
-

- CNV檢測於發育疾病
- SNP研究於慢性病(糖尿病等)
- 毒理學基因表現分析
- NGS:解鎖基因複雜性
-

- 全基因體定序(WGS)、外顯子定序(WES)
- 癌症液態切片追蹤突變
- 單細胞與微生物組分析
- 病原探索與新疾病發現
從桑格定序到次世代定序(NGS)的發展歷程,標誌著我們理解與應用人類基因體方式的重大轉變。如今,NGS 不僅實現了精準診斷,還推動個人化醫療的創新,並揭示過去無法想像的基因洞見。
對於臨床醫師、研究人員與醫療照護提供者而言,採用 NGS 技術意味著能夠更快速、更深入且更具行動力地掌握基因資訊,從而提升病患照護品質、加速科研進展,並強化全球公共衛生體系。
邁向全方位基因技術的趨勢
隨著基因組學的進步,對技術的期望也不斷提高。現代醫學不再滿足於僅識別少數幾個突變;它要求基因分析具備深度、廣度與整合性。
現代基因組學需要什麼?
- 深度
能夠檢測傳統方法可能漏掉的稀有和細微變異 - 廣度
分析整個基因組,而非僅限於編碼區域,以全面了解疾病的全貌 - 整合性
結合基因組、轉錄組和表觀基因組數據,揭示驅動疾病機制的複雜互動
為何次世代定序(NGS)是基因檢測的重大突破?
次世代定序(Next-Generation Sequencing, NGS)正引領精準醫療與基因檢測的全新變革,因為它能大規模解決臨床與研究的核心需求
前所未有的定序規模
NGS 能在單一實驗中定序整個基因體(genome)、外顯子體(exome)與轉錄體(transcriptome),非常適合進行全方位分析
極速處理
從過去需耗時數年,到如今僅需數天即可完成,大幅加快臨床決策,縮短診斷歷程
成本效益佳
基因體定序的費用已從數十億美元降至數千美元,讓高階診斷技術更普及、更可近
檢測面向廣泛
NGS 能一次性檢測多種基因變異,提升臨床與研究的檢測完整性:
- 單一核苷酸變異(SNVs)
- 插入與缺失變異(INDELs)
- 拷貝數變異(CNVs)
- 結構性重組
- 調控區域突變
基因體學入門|解碼健康、疾病與演化的關鍵
基因體學(Genomics)是研究基因體結構、功能與變異的科學,幫助我們深入了解人體健康、疾病機制與演化歷程
編碼區與非編碼區的差異
編碼DNA(僅佔全基因體的1–2%)
- 包含外顯子(exons),負責製造蛋白質
- 突變會直接影響胺基酸序列與蛋白功能
- 鐮刀型紅血球貧血,由β-珠蛋白基因單一鹼基變異引起
非編碼DNA(約佔98–99%)
- 調控基因表現、剪接與染色質結構
- 包含內含子(introns)、啟動子(promoters)、增強子(enhancers)與非編碼RNA
- 即使編碼序列正常,非編碼區突變也可能破壞調控機制,導致疾病
常見基因變異類型
單一核苷酸變異(SNVs)
類似句子中的「錯字」
- Single-base substitutions (e.g., A → T)
- 同義突變
- 不改變胺基酸(如 CGA → CGG,兩者皆為精胺酸)
- 非同義突變
- 改變胺基酸(如 GAG → GTG,導致鐮刀型紅血球貧血)
- 終止突變
- 產生提前終止密碼子,使蛋白質截短(如癌症中 TP53 突變)
插入/缺失變異(INDELs)
如句子中「遺漏或多出單字」
- 1~50個鹼基的插入或缺失
- 移碼突變(Frameshift)
- 若插入/缺失非3的倍數,將改變後續胺基酸序列 (例:BRCA1 中的4個鹼基缺失,會破壞抑癌功能)
拷貝數變異(CNVs)
如書中「重複或缺頁」
- 50個以上鹼基的片段複製或刪除
- 刪除
- 如 SMN1 缺失導致脊髓性肌肉萎縮症
- 重複
- 如 APP 基因重複會提高阿茲海默症風險
- 例:CCL3L1 拷貝數變異會影響 HIV 感染風險
為何漏掉基因變異可能代價高昂?
若基因檢測僅涵蓋編碼區域或解析度不足,可能錯過關鍵變異,造成:
錯失診斷、延誤治療

- 調控區域或結構變異可能未被偵測
- 罕見病或複雜病症患者可能多年沒診斷
治療決策失準

- 基因變異會影響藥物代謝與療效
- 例:未檢出 BRCA 突變,可能錯過預防性手術或標靶藥(如PARP抑制劑)機會
無法落實精準預防

- 高風險者可能錯失早期篩檢或個人化建議
- 包含生活調整、藥物介入與預防性處置
家族規劃風險增加

- 遺傳性疾病攜帶者若未被偵測
- 可能導致未預期的遺傳風險傳遞給下一代
✅ 用對視角,掌握全局
完整且整合的檢測方式——如次世代定序(NGS)——可協助臨床與研究單位:
- 全面捕捉重要基因變異
- 從整體基因體角度進行準確解讀
- 實現個人化診斷與治療
- 減少罕病患者的「診斷旅程」
當今的基因檢測現況
目前多數傳統基因檢測仍依賴晶片技術(Microarray),該技術針對已知的常見基因區域進行分析,雖能有效偵測大型變異與已知SNPs,卻在以下方面有所不足:
- 罕見或新型突變
- 結構變異(Structural Variations)
- 臨床重要的非編碼區域
這正是 生命藍圖™ 的突破之處。它採用次世代定序技術(NGS),實現更全面、深入且動態的DNA分析。
晶片技術 vs. 次世代定序(NGS):紙本地圖 vs. GPS 導航
晶片技術:紙本地圖
- 解析度有限
- 僅能檢視預設標記,忽略了許多關鍵變異
- 資訊靜態且過時
- 無法偵測目標區域外的新型突變
- 盲區多
- 難以處理如 PMS2 等複雜區域,常需補充 PCR 或 MLPA 等確認檢測
生命藍圖™ NGS:精準GPS導航

- 全面導航
- 採用 Cell3™ Target: Nexome,涵蓋編碼區與臨床重要非編碼區,檢出變異數比一般面板高30%
- 即時更新
- 同步偵測 SNVs、INDELs、CNVs,縮短報告時間並降低成本
- 動態精準
- 從微小突變(如 BRCA1 單一鹼基)到大規模變異(如 SMN1 缺失),一次偵測完成
生命藍圖™ 的關鍵優勢
傳統檢測猶如快速翻閱基因故事的一章章,而 生命藍圖™ 則是通讀全書,結合非編碼區、罕見突變與藥物反應基因,轉化為可實踐的健康建議
超越章節式分析
- 擴展型外顯子定序
- 涵蓋 51 Mb,包括難以捕捉的非編碼區(如癲癇、CYP2D6 藥物代謝)
- 多重變異偵測一次搞定
- 檢測 SNVs、INDELs、CNVs,無需多次測試
以精準為核心設計
- 專利探針設計
- 達成 94.18% 命中率,優於一般檢測的 85–93%
- 提供臨床關聯資訊
- 涵蓋 ACMG73(具臨床行動建議變異)、ClinVar(已知致病變異)、CPIC(藥物基因指引)等
晶片技術 vs. 次世代外顯子定序(WES)比較表
次世代定序技術(NGS),如全外顯子定序(WES),已徹底革新了基因檢測方式,提供遠超傳統晶片技術的深度洞察與精準結果。
| 功能項目 | 晶片技術(Microarray) | 進階外顯子定序 (Cell3™ Target: Nexome) |
| 覆蓋範圍 | 已知SNP與CNV | 蛋白質編碼區 + 非編碼區 |
| 準確度 | 小變異偵測較差 | 高精準度(SNV、INDEL、CNV) |
| 資科一致性 | 基本 | 先進探針確保一致性 |
| 偵測變異類型 | SNPs、CNVs | SNVs、INDELs、CNVs(一次完成) |
| 診斷率 | 較低 | 變異檢出率高出30% |
| 工作流程 | CNV需另行測試 | 一體化流程 |
| 成本效益 | 前期成本低 | 長期效益高,診斷率高,避免重複檢測 |
次世代定序的未來已來
次世代定序(NGS),尤其是結合 Cell3™ Target: Nexome 的進階 Whole Exome Sequencing(WES),正在重新定義基因檢測的可能性:
✅ 偵測更多基因變異
✅ 更快速且更準確的診斷
✅ 真正實現個人化醫療
無論是臨床治療、遺傳風險評估,還是家族健康規劃,生命藍圖™ 都能提供清晰可行的精準解答。
開啟個人化醫療的新篇章?
選擇 生命藍圖™,由 Cell3™ Target: Nexome 技術驅動,讓你自信邁入精準醫療的時代。
你的基因藍圖,你的未來,從今天啟程!