GALEAS™ Tumor
精密がん遺伝子解析ソリューション (NGS)
GALEAS™ Tumor は、SNV、CNV、INDEL、TMB、MSI、免疫腫瘍学的バイオマーカーなど、がん遺伝子解析に対応した最先端のNGSソリューションです
Advanced NGS-Based Identification of Tumor Mutations
GALEAS™ Tumor is a cutting-edge next-generation sequencing (NGS) solution designed to provide in-depth cancer genomic profiling. It enables the detection of clinically significant biomarkers, offering insights into tumor mutational burden (TMB), microsatellite instability (MSI), and structural variations.
GALEAS™ Tumor の主な特長
臨床的有用性と最新の内容
- 英国NHS National Genomic Test Directory、NCCN、FDA、ESMOの最新ガイドラインに準拠
- 519種類の遺伝子をカバー(SNV、INDEL、CNV、融合遺伝子を含む)
- TMBおよびMSIスコアにより免疫療法の効果予測を支援
- 64種の薬理ゲノムSNP、遺伝性および小児がんに関する情報、実体腫瘍におけるHLAプロファイリングを含む
高度な免疫腫瘍バイオマーカー解析
- TMBとMSIの統合解析により、腫瘍ゲノムの不安定性を評価
- 精密医療に基づいた免疫治療の予測精度を向上
高性能GALEAS™解析ソフトウェア
- クラウドベースのプラットフォームで、迅速かつ高精度な変異解析(SNV、INDEL、SV、CNV、TMB、MSI)を実現
- 1Mb以上の解像度によるCNV検出に対応
- パネルと同時に開発された、ユーザーフレンドリーなバイオインフォマティクスソリューション
GALEAS™ Tumor パネル設計
本次世代シーケンシング(NGS)ソリューションは、重要なドライバー変異および免疫腫瘍学に関連する主要なバイオマーカーを包括的にカバーしています:
- 個体識別用のトラッキングSNP
- 519種類の遺伝子にわたるSNV、CNV、INDELの解析
- 精度を高めたCNV検出のための強化バックボーン設計
- 神経膠腫に関連する1p/9q共欠失の包括的カバレッジ
- 以下を含む構造変異の検出に対応:ALK、BRAF、EGFR、FGFR2、FGFR3、NTRK1、NTRK2、RET、ROS1、TMPRSS2
- がん領域で臨床的意義を持つ64種類の薬理ゲノムSNP
- 実体腫瘍の特徴評価を目的としたHLA遺伝子プロファイリング
高精度なバリアント検出性能
SNVおよびINDELの検出精度
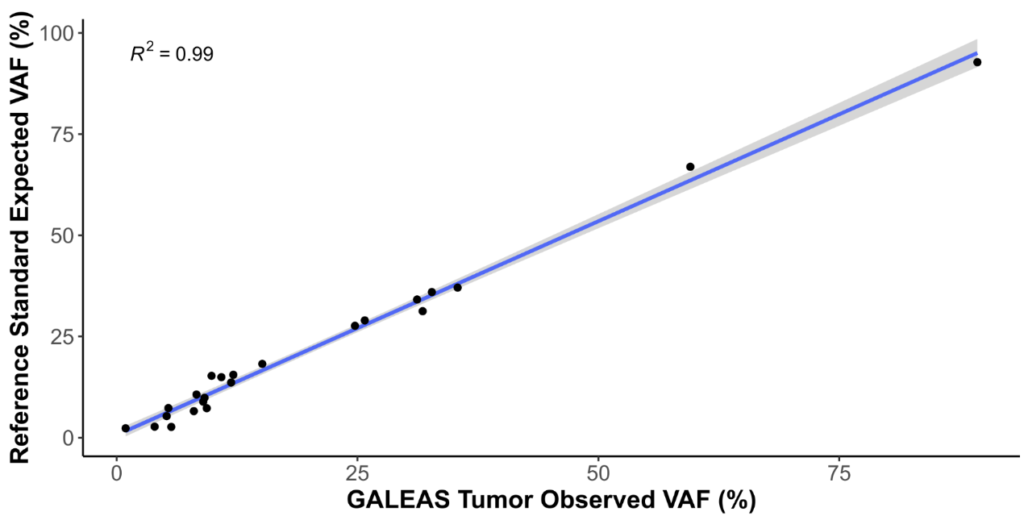
- FFPE参照サンプルを用いた検証により、500倍カバレッジで相関係数 R² = 0.99 を達成(図1)
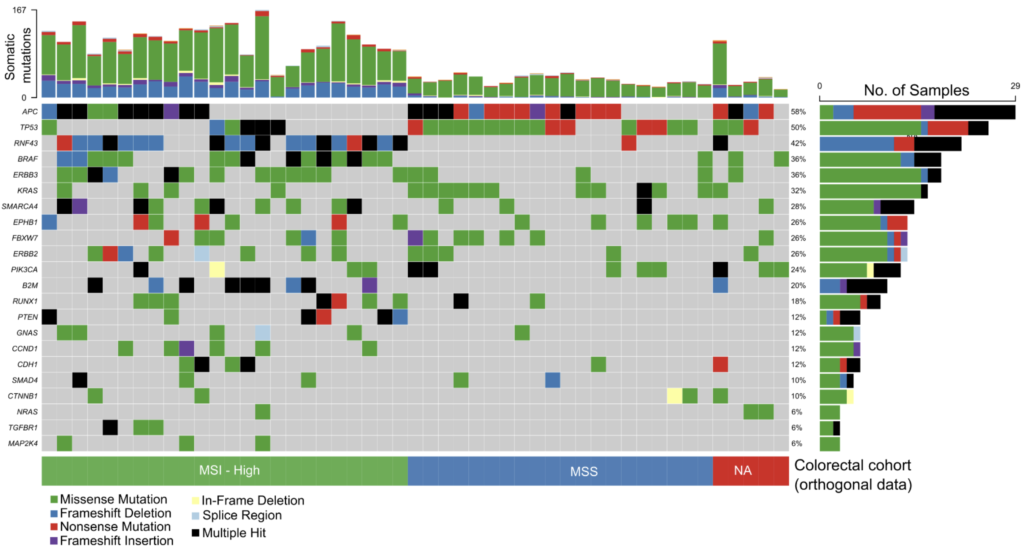
- 50件の大腸がん(CRC)サンプルにおいて体細胞変異の検出精度100%を実現(図2)
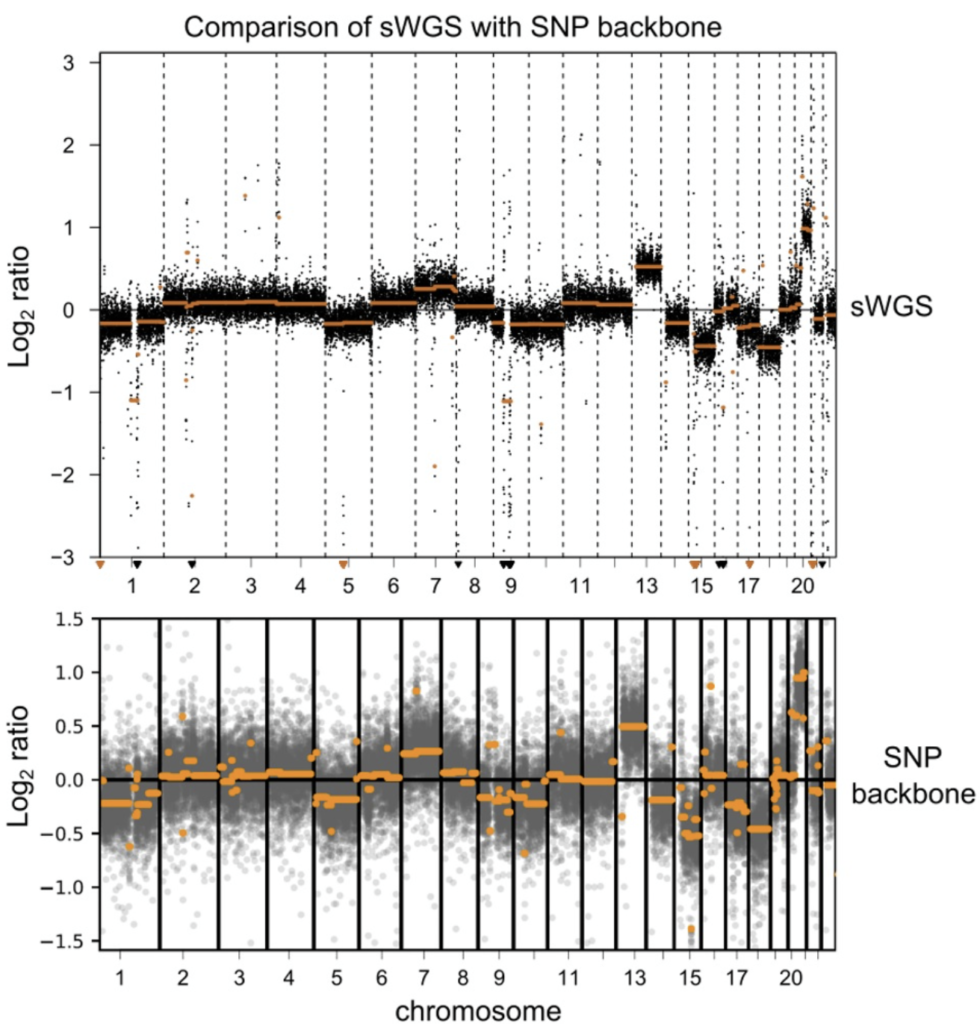
コピー数変異(CNV)検出
- 1Mbを超える解像度でのCNVコールに対応
- GALEAS™ TumorのCNVデータと浅層全ゲノムシーケンシング(sWGS)の結果との間に高い相関を確認(図3)
- EGFRおよびMET遺伝子のCNV変異(3、6、12コピー)検出を検証済み
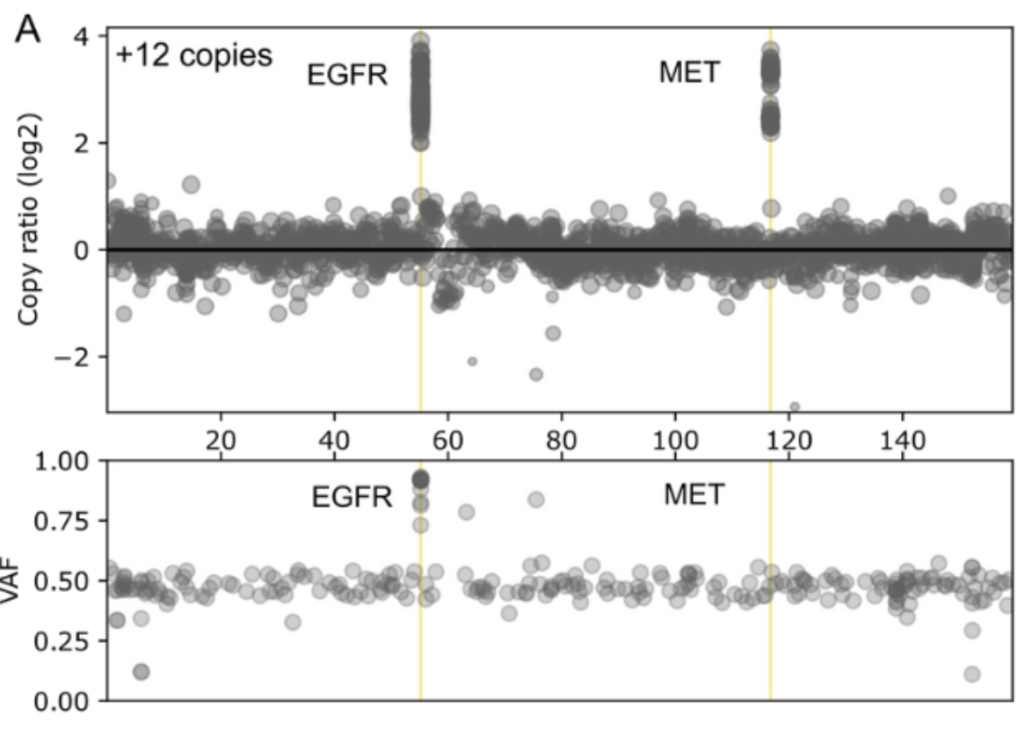
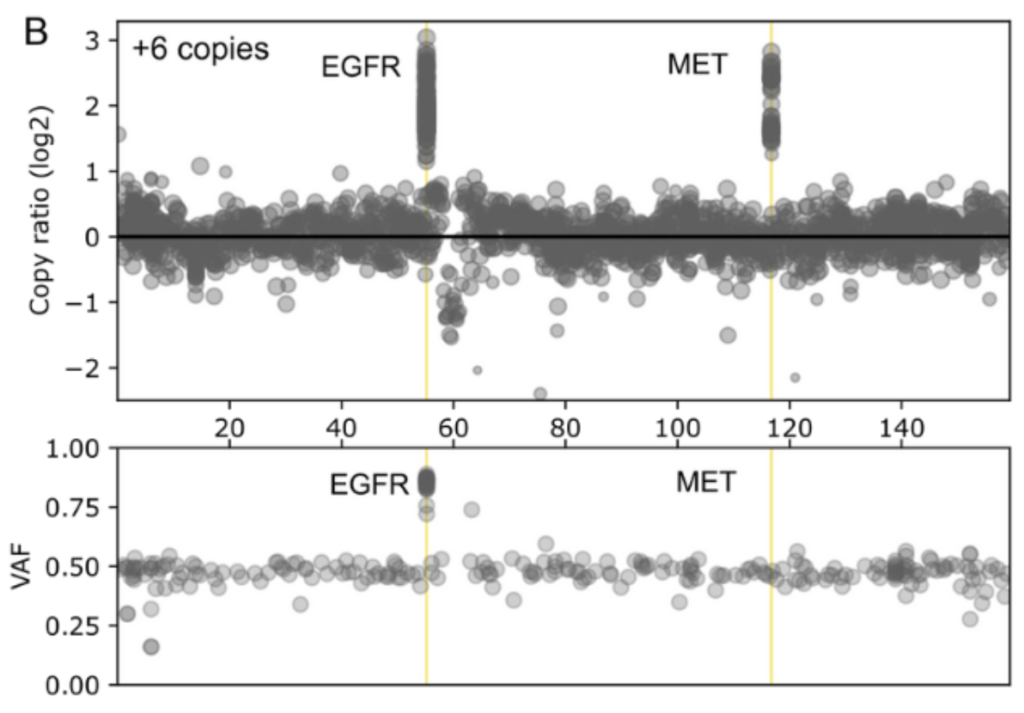
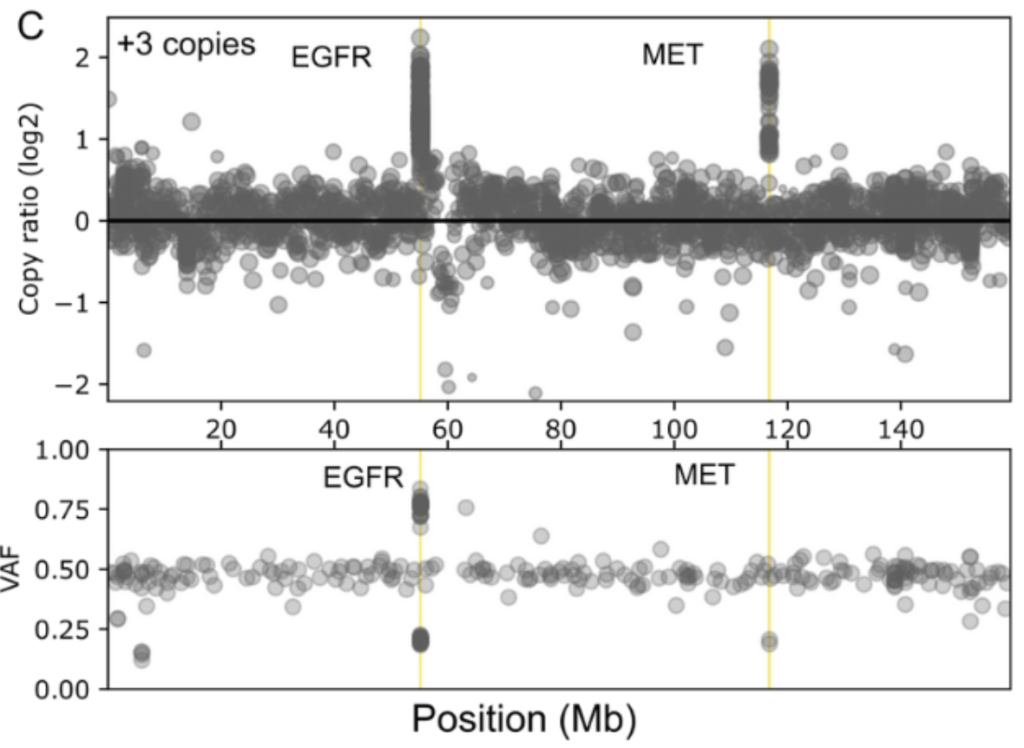
(A) 12コピー、(B) 6コピー、(C) 3コピーにおけるEGFRおよびMETの検出結果
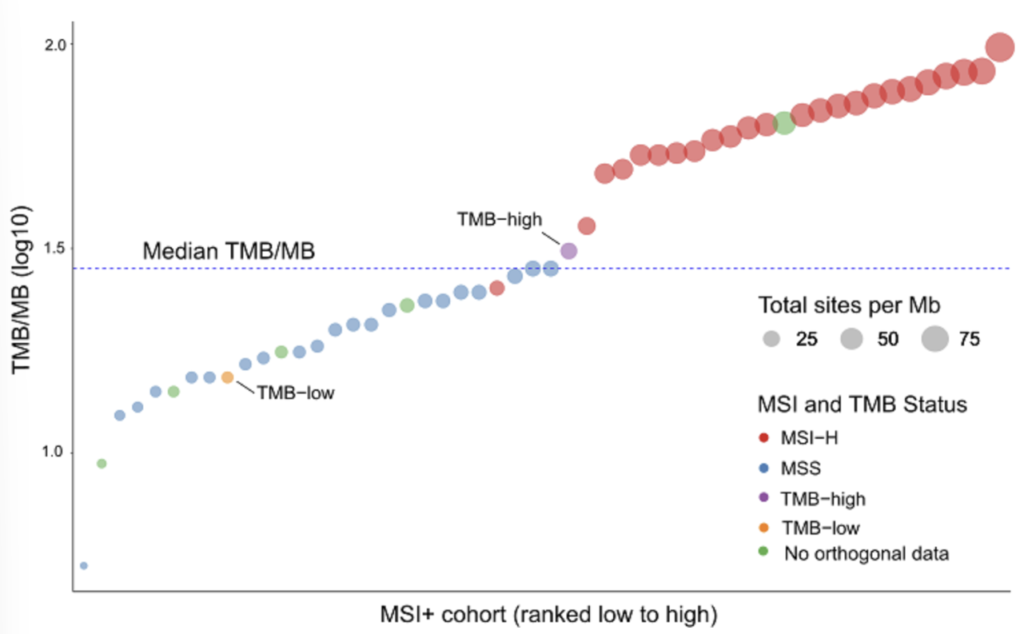
マイクロサテライト不安定性(MSI)スコアリング
- CRC FFPEサンプル24件中23件でMSI-Highステータスを正確に検出
- 正常、MSS(マイクロサテライト安定型)、MSI-H(高不安定型)の分類が可能
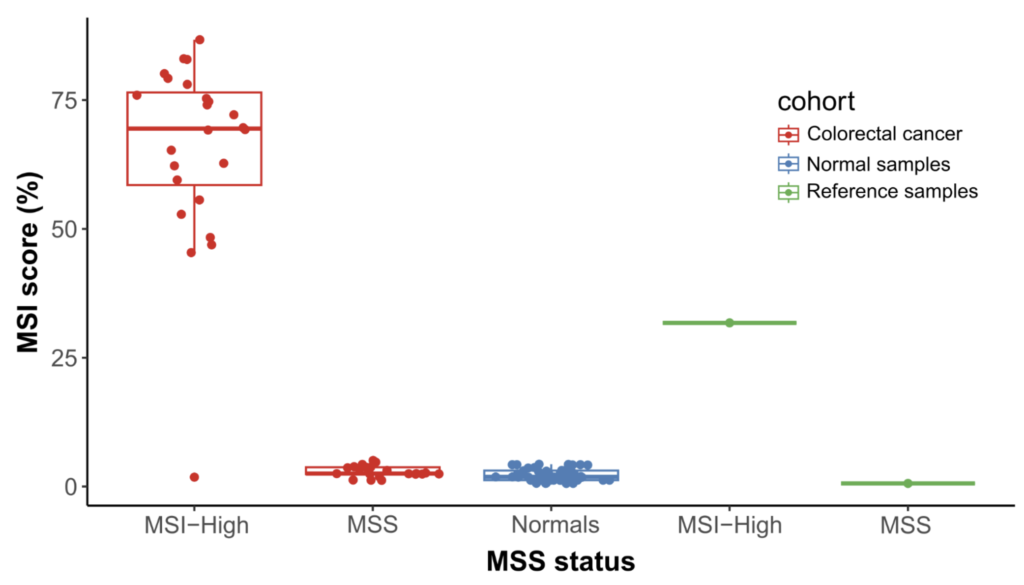
腫瘍変異負荷(TMB)解析
- CRCサンプルにおけるTMBスコアとMSIステータスの間に強い相関を確認
- 低変異量の小型パネルにおいても信頼性の高いTMBスコアリングを実現
- 高いオンターゲット率により、シーケンシング効率を最大化
- 臨床的に重要な遺伝子にわたって均一なカバレッジを実現
- 精密かつ再現性の高い変異検出 を可能にする設計
GALEAS™ 分析ソフトウェア
- GALEAS™ Tumor パネルのパフォーマンスを強化するために設計されています。
- TMB/MSI 分析を精度高く実施できる、効率的なバイオインフォマティクスワークフローを提供します。
GALEAS™ Tumor は、臨床的に検証された包括的な次世代シーケンシング(NGS)ソリューションで、単一のNGSワークフロー内で 519 種類の遺伝子に対する SNV、CNV、INDEL、TMB/MSI の分析を可能にします。
強化されたプローブ設計、包括的な遺伝子カバレッジ、均一なカバレッジ深度により、SNV、INDEL、SV、CNV の正確かつ敏感な検出が実現できます。GALEAS™ Analysis Software ソリューションと組み合わせることで、サンプル準備から分析までのシンプルで使いやすいワークフローを提供します。
References
- Ciriello G, Miller ML, Aksoy BA, Senbabaoglu Y, Schultz N, Sander C. Emerging landscape of oncogenic signatures across human cancers. Nature genetics. 2013;10):1127-33.
- The ICGC/TCGA PanCancer Analysis of Whole Genomes Consortium. Pan-cancer analysis of whole genomes. Na- ture. 2020: 82-93.
- Endris V, Buchhalter I, Allgäuer M, Rempel E, Lier A, Vol- ckmar AL, et al. Measurement of tumor mutational bur- den (TMB) in routine molecular diagnostics: in silico and real-life analysis of three larger gene panels. International journal of cancer. 2019;144(9):2303-12.
- Schrock AB, Ouyang C, Sandhu J, Sokol E, Jin D, Ross JS, et al. Tumor mutational burden is predictive of response to immune checkpoint inhibitors in MSI-high metastatic colorectal cancer. Annals of Oncology. 2019;30(7):1096-103.
製品仕様
| Parameters | Specification |
|---|---|
| Enrichment method | Hybridization and capture |
| Number of genes | 519 |
| Capture Panel Size | 3.74Mb |
| Sequencing platform | Illumina |
| Targets | Genes associated with common cancers |
| Variant types | SNVs, CNVs and indels |
| Input DNA requirements | 10ng-200ng |
| Sample type | gDNA from FFPE, frozen tissue or blood |
| Multiplexing guidance for sequencing | 25M reads per sample required to achieve 500x. This equates to 5Gb per sample |
注文情報
| Product Name | Catalogue No. |
|---|---|
| GALEAS™ Tumor Kit Frag A (96 samples) | NGS_GAL_TCP_FR_96_A |
| GALEAS™ Tumor Kit Frag B (96 samples) | NGS_GAL_TCP_FR_96_B |
| GALEAS™ Tumor Kit Frag C (96 samples) | NGS_GAL_TCP_FR_96_C |
| GALEAS™ Tumor Kit Frag D (96 samples) | NGS_GAL_TCP_FR_96_D |
| GALEAS™ Tumor Kit Frag (16 samples) | NGS_GAL_TCP_FR_16 |
- A:インデックス番号 1~96
- B:インデックス番号 97~192
- C:インデックス番号 193~288
- D: インデックス番号 289~384