GALEAS™ HereditaryPLUS
高精度な遺伝性腫瘍検出のための次世代検査パネル
GALEAS™ HereditaryPLUSは、146のがん関連遺伝子を解析し、SNV・INDEL・CNV・モザイク変異を高精度で検出。信頼性の高い遺伝子診断を実現します。
遺伝性癌リスクに対する先進的な遺伝子検査
GALEAS™ HereditaryPLUSは、遺伝性腫瘍の変異を精密に評価するために設計された最先端の遺伝性腫瘍パネルです。このパネルは、乳癌、前立腺癌、リンチ症候群、ウィルムス腫瘍など、さまざまな遺伝性癌に関連する146の臨床的に重要な遺伝子をカバーしています。
GALEAS™ HereditaryPLUS の主な特徴
包括的な遺伝子カバレッジ
- 遺伝性腫瘍に関連する146の高リスク遺伝子をターゲット
- 乳癌や前立腺癌などの一般的な遺伝性癌をカバー
- 嗜鉻細胞腫(pheochromocytoma)やウィルムス腫瘍(Wilms tumor)など、稀な遺伝性腫瘍にも対応
- BRCA1/2の5′ UTRやAPCプロモーターなど、臨床的に関連する非コード領域を網羅
表1:GALEAS™ HereditaryPLUSパネルに含まれる遺伝性癌リスクに関連する遺伝子のリスト
| Cancer type | Recommended genes for screening included in GALEASTM HereditaryPlus |
|---|---|
| Breast | ATM, BARD1, BRCA1, BRCA2, CDH1, CHEK2, NBN, NF1, PALB2, PTEN, STK11, TP53 |
| Colon | TAPC, AXIN2, BMPR1A, CHEK2, EPCAM, GREM1, MLH1, MSH2, MSH6, PMS2, MSH3, MUTYH, NTLH1, POLD1, POLE, PTEN, RNF43, SMAD4, STK11, TP53 |
| Renal | BAP1, FH, FLCN, MET, SDHB, VHL |
| Ovarian | ATM, BARD1, BRCA1, BRCA2, CDH1, CHEK2, NBN, NF1, PALB2, PTEN, SKT11, TP53, RAD51C, RAD51D |
| Prostate | ATM, BRCA1, BRCA2, CHEK2, MLH1, MSH2, MSH6, PALB2 |
| Gastric/GIST | CDH1, KIT, PDGFRA, SDHC, SDHD, SDHA |
| Brain | APC, ATM, MLH1, MSH2, MSH6, PMS2, TP53 |
| Sarcoma | EXT1, EXT2, MTAP, NF1, RECQL4, SQSTM1, TP53 |
| Paediatric* | CDKN1C, CTR9, REST, TRIM28, WT1 |
優れた精度と再現性
- 先進的なプローブ設計により、SNV、INDEL、CNVの検出感度が向上
- 高い均一性と包括的なカバレッジにより、診断の信頼性が向上
- 標準コントロールNA24385を用いた437のSNPの検証
表2:GALEAS™ HereditaryPLUSパネルにおけるNA24385標準サンプルでのSNVおよびINDELの再現性分析(4回の繰り返し実験)
| Recall | |
|---|---|
| SNV | 99.78% |
| Indel | 100% |
GALEAS™ 分析ソフトウェアによる最適化されたバイオインフォマティクス解析
- クラウドベースの解析パイプラインにより、高精度な変異判定を実現
- 付属の「ノーマルパネル」により、CNV検出精度を向上
- 主要な遺伝子(例:APC、TSC2)のモザイク型コピー数変異(Mosaic CNV)の検出をサポート
コピー数変異(CNV)の精密検出
- NIBSCリンチ症候群MLPA細胞株において100%の精度を達成
- 単一外顕子から全遺伝子にわたるCNVを高再現性で検出
- 主要な遺伝性癌遺伝子におけるモザイク型CNVの検出能力向上
表3:GALEAS™ HereditaryPLUSパネルを用いたNIBSCリファレンスコントロールでのCNV検出の召回率および精度統計
| CNV | Genotypic sex | CNV type | Detected |
|---|---|---|---|
| Copy normal | Male | Copy neutral | Yes |
| MSH2 deletion exons 1-6, heterozygous | Male | Multi-exon deletion | Yes |
| MSH2 deletion exon 7, heterozygous | 1 Male | Single exon deletion | Yes |
| MSH2 deletion exons 1-2, heterozygous | female | Multi-exon deletion | Yes |
| MSH2 deletion, exon 1, heterozygous | Male | Single exon deletion | Yes |
| MLH1 exon 13 amplification (3 or more copies) | Female | Multi-exon amplification | Yes |
優れたシーケンシングパフォーマンス
- 高いターゲット領域の読み取り率、低い重複率、均一なカバレッジ深度
- 99%のターゲット領域が30x以上のカバレッジでカバーされ、精度が保証される
- 競合製品に比べて、シーケンシングコストが増加せずに優れた性能を発揮
表4:GALEAS™ HereditaryPLUSと他の商業製品とのシーケンシングパフォーマンス比較
| Key Quality Indicator | GALEAS™ Hereditary Plus | Company I |
|---|---|---|
| Number of genes | 146 | 113 |
| Capture panel size (kb) | 809 kb | 403 kb |
| GB required for mean 100x coverage | 0.2 Gb | 0.12 Gb |
| Percentage coverage >30x | 99% | 96% |
| Percentage on or near bait | 81% | 61.51% |
| Percent duplication | 2.0% | 8.99% |
| SNV recall | 99.7% | 98.1% |
| INDELs recall | 100% | 97.2% |
臨床検証データ
SNV検出精度
- 臨床サンプルにおけるSNV召回率100%を達成
- 大きなINDELを含む多様な変異タイプを検出
表5:GALEAS™ HereditaryPLUSによる臨床サンプルでのSNV召回率
| ID | Gene | HGVS coding | HGVS protein | Genomic position |
|---|---|---|---|---|
| 22 | BRCA1 | c.1175_1214del | p.Leu392fs*5 | chr17:43094317 |
| 23 | BRCA1 | c.1175_1214del | p.Leu392fs*5 | chr17:43094317 |
| 64 | MSH2 | c.942+3A>T | P.? | chr2:47414421 |
| 65 | PMS2 | c.736_741delins TGTGTGTGAAG | p.(Pro246Cysfs*3) | chr7:5997389 |
| 66 | MLH1 | c.1946dupC | p.(Leu650Phefs*14) | chr3:37048561 |
| 67 | MSH2 | c.1213_1217dup | p.(Leu407Thrfs*7) | chr2:47429877 |
| 68 | MSH6 | c.3562_3563del | 1 p.(Ser1188Tyrfs*5) | chr2:47805623 |
コピー数変異(CNVs)検出
GALEAS™ HereditaryPLUSパネルとGALEAS™ 分析ソフトウェアを組み合わせることで、以下を実現:
- PMS2におけるCNV検出(図1C)
- 単一外顕子から全遺伝子までの広範囲なCNV検出
- APCおよびTSC2などの主要遺伝子におけるモザイク型CNVの検出(図1E、F)
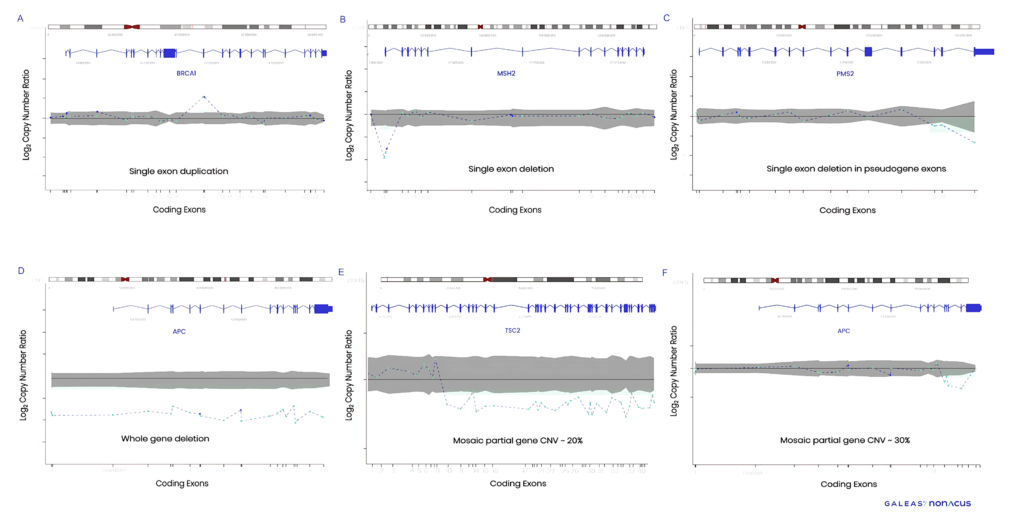
A)BRCA1 単一外顕子重複,B)MSH2 単一外顕子欠失,C)PMS2 偽遺伝子複数外顕子欠失,D)APC 全遺伝子欠失,E)TSC2 モザイク型部分遺伝子CNV -20%,F)APC モザイク型部分遺伝子CNV -30%
References
1. Ngeow J, Eng C. Precision medicine in heritable cancer: when somatic tumour testing and germline mutations meet. NPJ Genomic Medicine. 2016;(1):1-3.
製品仕様
| Parameters | Specification |
|---|---|
| Enrichment method | Hybridisation and capture |
| Number of genes | 146 |
| Capture Panel Size | 809 Kb |
| Sequencing platform | Illumina |
| Targets | Genes associated with hereditary cancer |
| Variant types | SNVs, INDELs and CNVs |
| Sample type | gDNA from blood or saliva |
| Input DNA requirements | 10-200 ng |
| Multiplexing guidance for sequencing | 1 million reads per sample required to achieve 100×. This equates to 0.2 Gb per sample |
注文情報
| Product | Catalog No. |
|---|---|
| GALEAS™ HereditaryPlus – 16 samples (with enzymatic fragmentation for gDNA or FFPE) | NGS_GAL_HCP_FR_16 |
| GALEAS™ HereditaryPlus – 96 samples (with enzymatic fragmentation for gDNA or FFPE) | NGS_GAL_HCP_FR_96_A/B/C/D* |
- A:インデックス番号 1~96
- B:インデックス番号 97~192
- C:インデックス番号 193~288
- D: インデックス番号 289~384
* サンプルのマルチプレックス化に柔軟性を持たせるため、96検体用キットにはアダプタープレートを提供しています。