GALEAS™ Hereditary Plus
遺伝性腫瘍パネル
GALEAS™ Hereditary Plusの特長
臨床的に重要な領域を強化
乳がん、前立腺がん、リンチ症候群、ウィルムス腫瘍など、遺伝性腫瘍に関連する146遺伝子の臨床的に重要な領域をキュレーションしてカバー
優れた精度と再現率
強化されたプローブ設計、包括的なカバレッジ、高いカバレッジと均一性により、SNV、INDELおよびCNVの正確かつ高感度な検出が可能
GALEAS™解析ソフトウェアがサポート
専用に最適化されたクラウドベースのバイオインフォマティクスパイプラインは、遺伝性腫瘍に関連するさまざまなバリアントに対応し、正確な結果を提供
パネル設計
遺伝性腫瘍に関連する146遺伝子をターゲットに設計されています。これらの遺伝子は、乳がんや前立腺がんなどの遺伝性腫瘍だけでなく、褐色細胞腫やウィルムス腫瘍などの小児がんといったまれな遺伝性腫瘍も対象としています。(表1)
また、臨床的に重要な領域がカバーされるよう慎重にキュレーションされており、BRCA1/2の5‘ UTRやAPCプロモーターなどの非コード領域も含まれています。
専用のGALEAS™解析ソフトウェアと組み合わせることで、SNV、INDEL、CNVを高感度かつ高特異度で検出することが可能です。
表1: GALEAS™ HereditaryPlusに含まれる遺伝性腫瘍の主要ガイドラインで推奨される遺伝子
| Cancer type | Recommended genes for screening included in GALEAS™ Hereditary Plus |
|---|---|
| Breast | ATM, BARD1, BRCA1, BRCA2, CDH1, CHEK2, NBN, NF1, PALB2, PTEN, STK11, TP53 |
| Colon | TAPC, AXIN2, BMPR1A, CHEK2, EPCAM, GREM1, MLH1, MSH2, MSH6, PMS2, MSH3, MUTYH, NTLH1, POLD1, POLE, PTEN, RNF43, SMAD4, STK11, TP53 |
| Renal | BAP1, FH, FLCN, MET, SDHB, VHL |
| Ovarian | ATM, BARD1, BRCA1, BRCA2, CDH1, CHEK2, NBN, NF1, PALB2, PTEN, SKT11, TP53, RAD51C, RAD51D |
| Prostate | ATM, BRCA1, BRCA2, CHEK2, MLH1, MSH2, MSH6, PALB2 |
| Gastric/GIST | CDH1, KIT, PDGFRA, SDHC, SDHD, SDHA |
| Brain | APC, ATM, MLH1, MSH2, MSH6, PMS2, TP53 |
| Sarcoma | EXT1, EXT2, MTAP, NF1, RECQL4, SQSTM1, TP53 |
| Paediatric* | CDKN1C, CTR9, REST, TRIM28, WT1 |
パネルの検証
優れた精度と再現率により、SNVおよびINDELを正確に検出します。市販の標準コントロールNA24385を使用し、437のSNPにおいて検証されました。(表2)
表2:GALEAS™ Hereditary PlusのSNVおよびインデルにおける再現率
(標準コントロールNA24385の4つの複製での結果)
| Recall | |
|---|---|
| SNV | 99.78% |
| Indel | 100% |
コピー数変異(CNV)の検出において 高い信頼性を確保
CNVジェノタイピングの感度を評価するために、NIBSCリンチ症候群MLPA細胞株を使用して解析を行いました。すべてのCNVが100%の再現率と精度で検出されました。(表3)
表3
| CNV | Genotypic sex | CNV type | Detected |
|---|---|---|---|
| Copy normal | Male | Copy neutral | Yes |
| MSH2 deletion exons 1-6, heterozygous | Male | Multi-exon deletion | Yes |
| MSH2 deletion exon 7, heterozygous | 1 Male | Single exon deletion | Yes |
| MSH2 deletion exons 1-2, heterozygous | female | Multi-exon deletion | Yes |
| MSH2 deletion, exon 1, heterozygous | Male | Single exon deletion | Yes |
| MLH1 exon 13 amplification (3 or more copies) | Female | Multi-exon amplification | Yes |
パネルの性能
高いオンターゲット率、低い重複率、そしてより一貫した垂直カバレッジを提供し、ターゲットの99%が30x以上でカバーされています。(表4)
この優れた技術性能により、CNVを含む遺伝性がんに関連するより多くのバリアントで高い再現率と精度を実現し、主要な競合他社と比較してもシーケンシングコストを大幅に増加させることなく優位性を発揮します。
表4:GALEAS™ Hereditary Plusのシーケンシング指標
| Key Quality Indicator | GALEAS™ Hereditary Plus | Company I |
|---|---|---|
| Number of genes | 146 | 113 |
| Capture panel size (kb) | 809 kb | 403 kb |
| GB required for mean 100x coverage | 0.2 Gb | 0.12 Gb |
| Percentage coverage >30x | 99% | 96% |
| Percentage on or near bait | 81% | 61.51% |
| Percent duplication | 2.0% | 8.99% |
| SNV recall | 99.7% | 98.1% |
| INDELs recall | 100% | 97.2% |
臨床検証
臨床的有用性は、64例の患者の血液サンプルから得られたgDNAを含む、協力者のサンプルを使用して評価されました。
SNVの再現率と精度
臨床サンプルにおけるSNVの再現率は100%であり、小さなものから大きなもの(>10 bpのINDEL)まで、さまざまな変異タイプにわたって確認されました。(表5)
表5:GALEAS Hereditary Plusにおける臨床サンプルでのSNV再現率
| ID | Gene | HGVS coding | HGVS protein | Genomic position |
|---|---|---|---|---|
| 22 | BRCA1 | c.1175_1214del | p.Leu392fs*5 | chr17:43094317 |
| 23 | BRCA1 | c.1175_1214del | p.Leu392fs*5 | chr17:43094317 |
| 64 | MSH2 | c.942+3A>T | P.? | chr2:47414421 |
| 65 | PMS2 | c.736_741delins TGTGTGTGAAG | p.(Pro246Cysfs*3) | chr7:5997389 |
| 66 | MLH1 | c.1946dupC | p.(Leu650Phefs*14) | chr3:37048561 |
| 67 | MSH2 | c.1213_1217dup | p.(Leu407Thrfs*7) | chr2:47429877 |
| 68 | MSH6 | c.3562_3563del | 1 p.(Ser1188Tyrfs*5) | chr2:47805623 |
コピー数変異(CNV)
専門家が構築した CNV プローブ設計と最適化されたバイオインフォマティクスパイプラインの組み合わせにより、次のことが可能になります。
•PMS2におけるCNV(図1C)
•単一エクソンから遺伝子全体まで、幅広いCNVを検出
•APCやTSC2などの重要な遺伝子におけるモザイク型のコピー数変異(図1EおよびF)
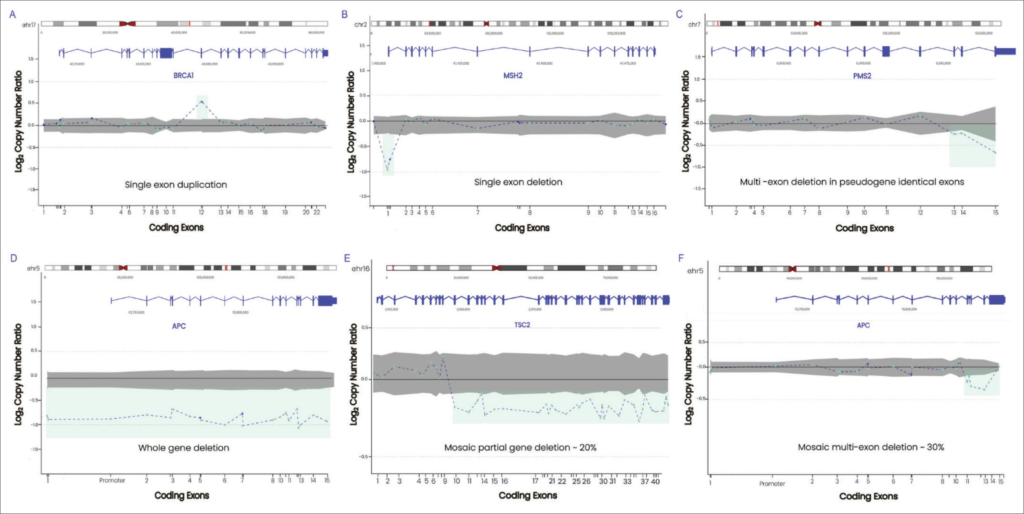
A) BRCA1 単一エクソン重複、B) MSH2 単一エクソン欠失、C) 偽遺伝子におけるPMS2 複数エクソン欠失、D) APC 全遺伝子欠失、E) TSC2 モザイク部分遺伝子 CNV -20%、F) APC モザイク部分遺伝子 CNV – 30%
GALEAS™解析ソフトウェア
最適化されたバイオインフォマティクスパイプラインのクラウドベースのセットで、単一エクソンから全遺伝子にわたるSNV、INDEL、および広範囲のCNVを正確にコールします。
Panel of Normals
バックグラウンドノイズを最小限に抑え、CNVコールを改善するために、GALEAS™解析ソフトウェアは組み込みのPanel of Normalsを活用しています。このデータセットは、臨床的に正常な大規模なサンプルコホートを使用したCNVをコールするための基準値を提供し、CNVコールの精度を大幅に向上させます。
まとめ
GALEAS™ Hereditary Plusは、遺伝性腫瘍に関連する遺伝子の解析に特化した、専門的にキュレーションされた製品です。強化されたプローブ設計、包括的なカバレッジ、高いカバレッジと均一性により、SNV、INDELおよびCNVの正確かつ高感度な検出が可能です。これにGALEAS™解析ソフトウェアを組み合わせることで、シンプルで簡単なサンプルから解析までのワークフローを提供します。
References
1. Ngeow J, Eng C. Precision medicine in heritable cancer: when somatic tumour testing and germline mutations meet. NPJ Genomic Medicine. 2016;(1):1-3.
製品仕様
| Enrichment method | Hybridisation and capture |
|---|---|
| Number of genes | 146 |
| Capture Panel Size | 809 Kb |
| Sequencing platform | Illumina |
| Targets | Genes associated with hereditary cancer |
| Variant types | SNVs, INDELs and CNVs |
| Sample type | gDNA from blood or saliva |
| Input DNA requirements | 10 ng – 200 ng |
| Multiplexing guidance for sequencing | 500K reads per sample required to achieve 100×. This equates to 0.2 Gb per sample |
注文情報
| Product | Catalog No. |
|---|---|
| GALEAS™ HereditaryPlus – 16 samples (with enzymatic fragmentation for gDNA or FFPE) | NGS_GAL_HCP_FR_16 |
| GALEAS™ HereditaryPlus – 96 samples (with enzymatic fragmentation for gDNA or FFPE) | NGS_GAL_HCP_FR_96_A/B/C/D* |
- A:インデックス番号 1~96
- B:インデックス番号 97~192
- C:インデックス番号 193~288
- D: インデックス番号 289~384
* サンプルのマルチプレックス化に柔軟性を持たせるため、96検体用キットにはアダプタープレートを提供しています。