Cell3™ Target: Nexome
臨床的に重要な領域を強化
単一のアッセイでSNV、INDEL、CNVを検出
Nexome vs. 従来製品
Cell3™ Target: Nexomeは、染色体マイクロアレイ(CMA)、多重リガンド依存プローブ増幅法(MLPA)、蛍光in situハイブリダイゼーション(FISH)といった複数の解析を回避することで、診断プロセスを効率化します。
これにより、以下の利点が得られます:
コスト削減
従来のワークフローで必要とされる複数のアッセイを行うことに比べて、コスト効率の高いソリューションを提供
迅速な結果の提供
複数のアッセイを統合することで、より早く結果を得ることができ、迅速かつ的確な臨床判断が可能
最小限のサンプル量
少量のサンプルで済むため、サンプルが限られている状況でも解析が可能
臨床的に重要な遺伝子を網羅
Cell3™ Target: Nexomeは、多くのエクソーム製品よりも広い領域をカバーし、ヒトゲノムのタンパク質をコードする領域だけでなく、臨床的に重要な非コード領域も対象としています。また、既知の遺伝子およびエクソンレベルの再構成が確認されている領域におけるCNV検出を可能にするプローブの強化も行われています。CCDS、GENCODE、RefSeq、ACMG73データベースに対して優れた網羅範囲を提供し、臨床的に重要な遺伝子やCNV検出に対応するプローブを強化しています。(図1)
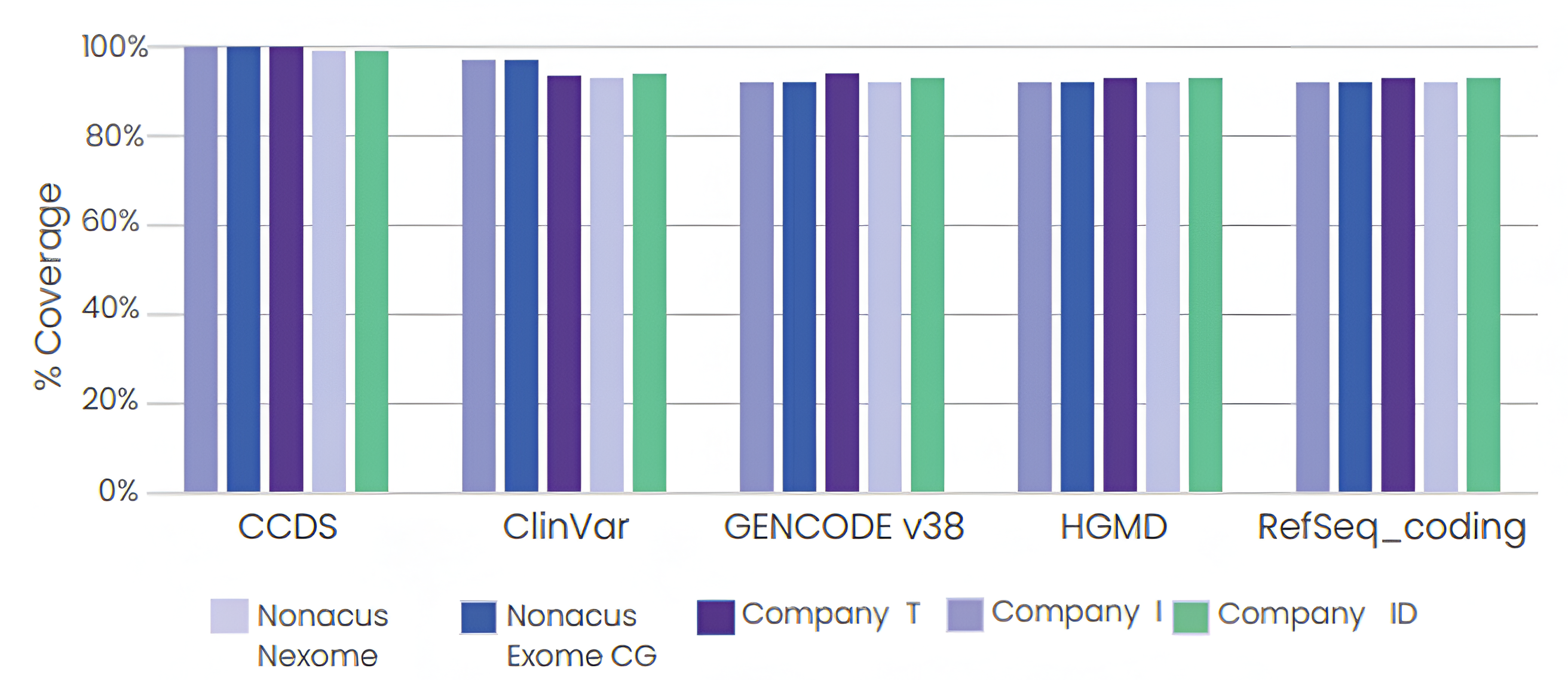
さまざまなデータベースに対する他のエクソーム製品とのカバレッジの比較
臨床的に強化された領域
- 市販のキット(例:MLPA)でターゲットとされているエクソンレベルの欠失や重複
- 出生前診断のための胎児異常に関連する遺伝子
- てんかんの診断を強化するため、早期乳児てんかん性脳症(EIEE)に関連する転写産物や追加エクソン
- OMIMに登録されている疾患に関連する4090の遺伝子に対応するRefSeq転写産物、プロモーター領域、5’UTRと3’UTR
- 非コード領域にある疾患の原因となるバリアント
- 薬物応答予測のための薬理ゲノミクス(PGx)マーカー
SNVおよびINDELの精度とリコール率
Cell3™ Target: Nexomeは、HG001ヒトゲノム標準リファレンスに存在する真のバリアントを他のエクソーム製品よりも多く検出します。また、SNVおよびINDELの両方において、優れた精度を維持しながら、同等のリコール率を確保しています。(図2a, 2b, 3a, 3b)
SNV
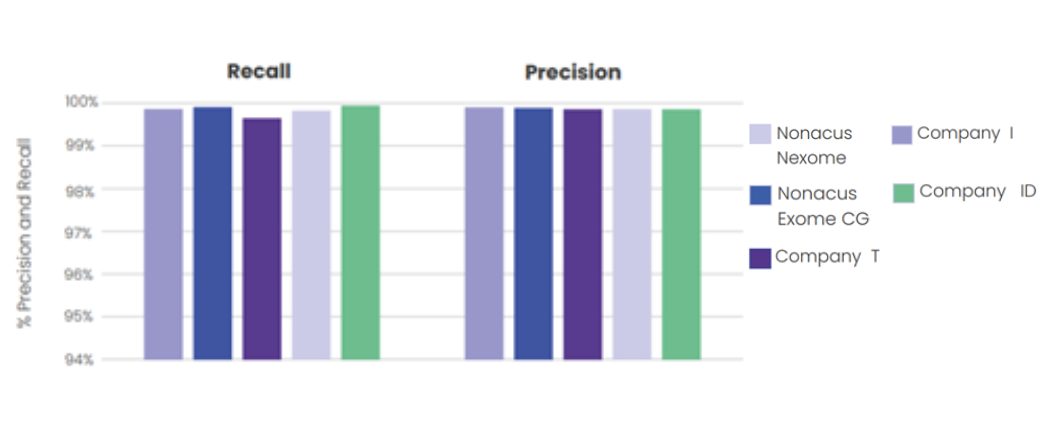
Nexomeおよび他のエクソーム製品で検出された
SNVの精度とリコール率
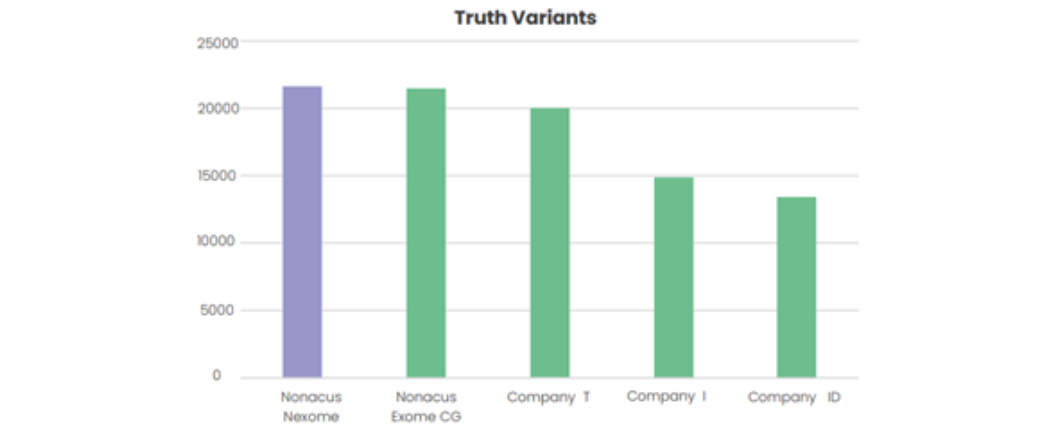
Nexomeおよび他のエクソーム製品で検出された
真のバリアント(SNV)の総数
INDELS
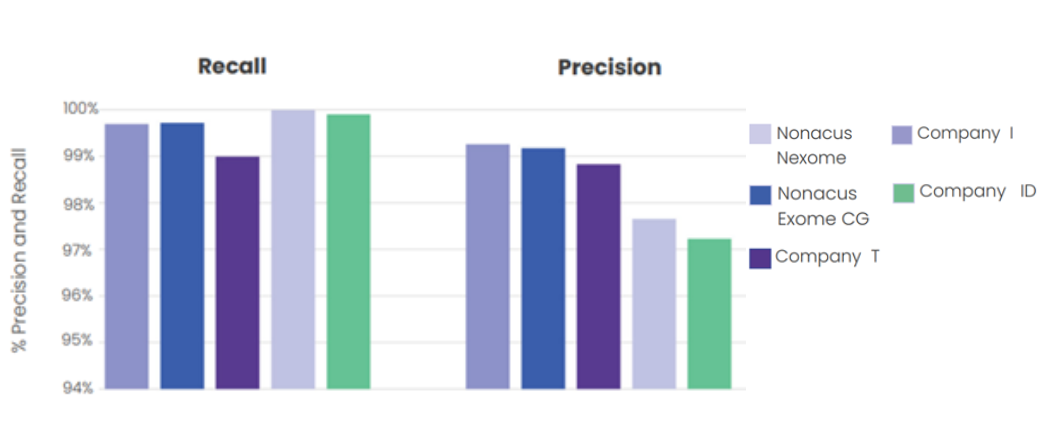
Nexomeおよび他のエクソーム製品で検出された
INDELの精度とリコール率
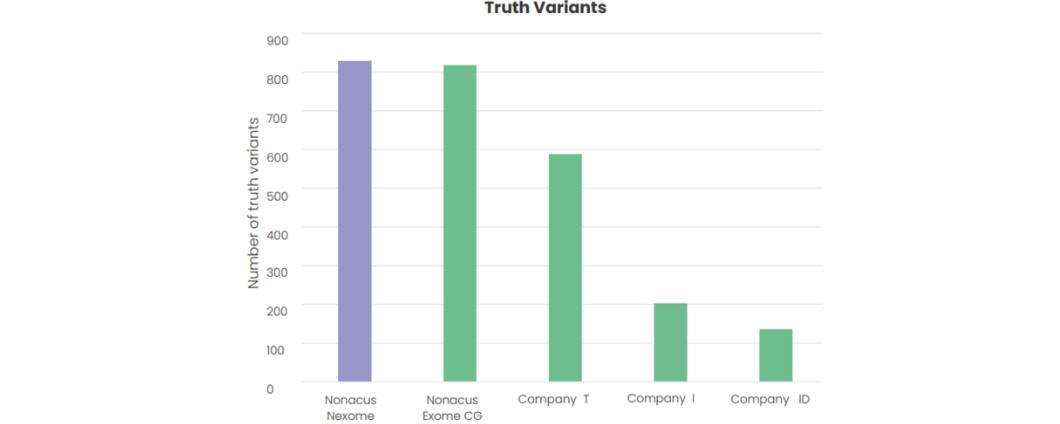
Nexomeおよび他のエクソーム製品で検出された
真のバリアント(INDEL)の総数
より多くの情報を提供しながら、
シーケンシングコストや追加のアッセイを増やさない
- ヒトゲノムの51Mb超をターゲット
- 他のエクソーム製品と比べ、最大30%多くのバリアントを呼び出し、幅広くCNVを検出できるように設計
- 優れたプローブ設計と性能により、他のエクソーム製品と比較して、同等または少ないシーケンス量(6.63 Gb)で、平均カバレッジ100×を達成(表1)
表1
| Panel Size (Mb) | Percentage target covered at 1x | Gb Required for mean 100x coverage | Percent Bases on or near bait | |
|---|---|---|---|---|
| Nexome | 51.90 | 98.78% | 6.63 | 94.18% |
| Exome CG | 51.60 | 98.78% | 6.57 | 94.07% |
| Company T | 36.70 | 97.42% | 6.85 | 85.89% |
| Company I | 45.20 | 98.15% | 7.16 | 86.18% |
| Company I.D | 34.10 | 98.49% | 6.04 | 93.09% |
精密なCNV検出
コピー数変異(CNV)は、疾患に関連するバリアントの約10%を占め、神経発達障害を持つ人の約10〜20%において特定されています。
- コピー数変異(CNV)は、疾患に関連するバリアントの約10%を占め、神経発達障害を持つ人の約10〜20%において特定されています。
- 遺伝子およびエクソンレベルの再構成が確認されている領域で、優れたCNVの検出性能を発揮するよう設計
- 数エクソンから複数の連続する遺伝子(約100 bpから40 Mbの範囲)にわたるCNVの検出が可能
- MLPAまたはCMAでテストされた既知のCNVイベントを持つサンプルを使用して評価し、50 bp(単一エクソン)から42 MbまでのCNV変異を検出することを確認(表1aおよび1b)
表1a: MLPAで確認されたCNVの検出
| Affected Gene | CNV region | CNV size (bp) | CNV exons | CNV type | Bayes factor |
|---|---|---|---|---|---|
| FBN1 | exons 29-65 | 74632 | 37 | deletion | 320.0 |
| BRCA1 | exons 1-23 | 77841 | 24 | deletion | 190.0 |
| FBN1 | exons 1-17 | 142063 | 18 | deletion | 300.0 |
| BRCA1 | exons 1-17 | 57876 | 18 | deletion | 200.0 |
| BRCA1 | exons 8-13 | 17956 | 6 | deletion | 40.4 |
| BRCA1 | exons 8-13 | 17956 | 6 | deletion | 82.4 |
| BRCA2 | exons 5-7 | 513 | 3 | deletion | 22.1 |
| NSD1 | exons 7-9 | 6034 | 3 | deletion | 34.5 |
| FBN1 | exons 60-62 | 3934 | 3 | deletion | 32.8 |
| NSD1 | exons 1-3 | 58095 | 3 | deletion | 54.8 |
| BRCA2 | exons 1-2 | 1054 | 2 | deletion | 28.3 |
| BRCA1 | exons 7-8 | 311 | 2 | deletion | 4.7 |
| BRCA1 | exons 8-9 | 1444 | 2 | deletion | 7.5 |
| BRCA1 | exon 16 | 211 | 1 | deletion | 14.5 |
| BRCA1 | exon 20 | 84 | 1 | deletion | 9.4 |
表1b: CMA で確認された複数遺伝子 CNV の検出
| CNV region | CNV size (Mb) | CNV genes | CNV type | Bayes factor |
|---|---|---|---|---|
| 13q14.2q32.1 | 42.0 | 367 | deletion | 2410 |
| 4p16.3p15.2 | 22.9 | 339 | deletion | 4620 |
| 20q11.22q13.12 | 11.3 | 244 | deletion | 7000 |
| 7p14.1p11.2 | 15.9 | 182 | deletion | 5040 |
| 1p36.32 | 3.7 | 140 | deletion | 2710 |
| 22q11.21 | 2.0 | 83 | deletion | 2890 |
| 8q23.1q24.12 | 11.8 | 71 | deletion | 1330 |
| 22q11.21 | 2.2 | 64 | duplication | 1430 |
| 11p12p11.2 | 2.3 | 54 | deletion | 1240 |
| 7q11.23 | 1.4 | 38 | deletion | 2080 |
| 15q11.2 | 0.9 | 31 | deletion | 494 |
| 17p12 | 1.3 | 24 | deletion | 275 |
| 14q22.1 | 0.7 | 20 | deletion | 508 |
| 15q11.2 | 0.5 | 4 | duplication | 370 |
| 13q12.11 | 0.2 | 2 | deletion | 75 |
シンプルで自動化可能なプロトコルの利点
- Cell3™ Target: Nexomeのキットには、ライブラリ調整、ハイブリダイゼーションとキャプチャに必要な試薬がすべて含まれています。
- Cell3™ Target: Nexomeのワークフローはシンプルで簡単に設計されており、所要時間は 10 時間未満、実作業時間は 2 時間未満です。
- 必要な DNA はわずか 1 ng
- 複数の停止ポイントが設計されており、ラボ内での処理において柔軟性を持たせることができます。
- ライブラリ調製は手動で行うことも、自動化することも可能で、最大96検体を1回のバッチで処理できます。
- インデックスは最大384検体まで対応しており、ハイスループットなラボにおいて柔軟なバッチサイズを可能にし、すべてのIlluminaシーケンサーに対してスケーラビリティを提供します。
References
1.McKusick V. Online Mendelian Inheritance in Man, OMIM™. McKusick-Nathans Institute for Genetic Medicine, Johns Hopkins University (Baltimore, MD) and National Center for Biotechnology Information, National Library of Medicine (Bethesda, MD), 2000. Accessed October 10, 2023. https://omim. org. 2009. Full article
2.Smedley D, Schubach M, Jacobsen JO, Köhler S, Zemojtel T, Spielmann M, et al. A whole-genome analysis framework for effective identification of pathogenic regulatory variants in Mendelian disease. The American Journal of Human Genetics. 2016; 99(3):595-606. Full article
3. Landrum MJ, Lee JM, Benson M, Brown GR, Chao C, Chitipiralla S, et al. ClinVar: improving access to variant interpretations and supporting evidence. Nucleic Acids Research. 2018;46(D1):D1062-7.. Full article
製品仕様
| Enrichment method | Hybridisation and capture |
| Capture panel Size | 51.9 Mb |
| Sequencing platform | Illumina |
| Targets | Clinically relevant genes |
| Variant types | SNVs, indels and CNVs |
| Sample type | gDNA from blood, saliva, amniotic fluid, tissue or FFPE, cfDNA |
| Input DNA requirements | 1-1000 ng |
| Expected percentage duplication | ~5-6% |
| Expected percentage on target (150 bp padding) | ~95% |
| Gb required for mean 100× coverage | 6.63 |
| Multiplex capability | 384 |
注文情報
Cell3™ Targetパネルは、2種類のライブラリ調製キットをご用意しています。
- フラグメンテーション対応キット:DNA(Genomic、FF、FFPE)サンプルに使用します。
- ノンフラグメンテーション対応キット:cfDNAに使用します。
| Product | Catalog No. |
|---|---|
| Cell3™ Target: Nexome, Frag 16 samples | NGS_C3T_NEX_FR_16 |
| Cell3™ Target: Nexome, Frag 96 samples | NGS_C3T_NEX_FR_96_A/B/C/D* |
| Cell3™ Target: Nexome, Non Frag 16 samples | NGS_C3T_NEX_NF_16 |
| Cell3™ Target: Nexome, Non Frag 96 samples | NGS_C3T_NEX_NF_96_A/B/C/D* |
- A:インデックス番号 1~96
- B:インデックス番号 97~192
- C:インデックス番号 193~288
- D: インデックス番号 289~384
* サンプルのマルチプレックス化に柔軟性を持たせるため、96検体用キットにはアダプタープレートを提供しています。